2017.02.01.5
Files > Volume 2 > Vol 2 No 1 2017 > Investigaciones
INVESTIGACIÓN
Identificación y cuantificación de Salmonella sp. y ADNr 16S bacteriano mediante PCR en tiempo real en muestras de alimentos
Identification and quantification of Salmonella sp. and bacterial DNAr 16S by Real-Time PCR in food samples
Viviana Chiluisa-Utreras 1; Andrea Echeverría 1
Available from: http://dx.doi.org/10.21931/RB/2017.02.01.5
_______________________________________________________________________________________________________________________
Resumen
Utilizando la técnica PCR en Tiempo Real, que es más sensible, específica y rápida que las técnicas convencionales, se ha logrado la determinación de algunos patógenos alimentarios de forma inmediata como por ejemplo: Salmonella sp.; que en pequeñas cantidades es capaz de causar enfermedades gastrointestinales resultando riesgoso para la salud del consumidor, la ingesta de alimentos sin control sanitario. La presente investigación se realizó con el objetivo principal de identificar y cuantificar mediante PCR en Tiempo Real la presencia de Salmonella sp. y ADNr 16S bacteriano en alimentos de establecimientos cercanos a la Universidad Politécnica Salesiana. Se tomaron muestras de diez locales y se analizaron por triplicado para posteriormente aplicar los análisis estadísticos pertinentes. Como resultado, utilizando la prueba estadística no paramétrica Kruskall-Wallis se detectó en el 100% de las muestras la presencia de la secuencia de ADNr 16S, que mostró significancia estadística; mientras que la evaluación de la secuencia SPI-1, un gen de virulencia para Salmonella sp., el 6,7% de casos positivos y se reveló que no existe diferencia significativa. Mediante las curvas estándar de cada secuencia, se logró cuantificar la carga bacteriana para 16S en un promedio de 51 ug/mL y para el caso de Salmonella sp. una concentración en el rango de 0,766 a 0,0170 ug/mL.
Palabras clave: Salmonella sp., ADNr 16S bacteriano, PCR en Tiempo Real, alimentos.
_______________________________________________________________________________________________________________________
Abstract
By using the real-time PCR technique, which is more sensitive, specific and faster than conventional techniques, the determination of some food pathogenic microorganisms has been achieved immediately, as in the case of Salmonella sp.; which in minimal amounts is capable of causing gastrointestinal diseases resulting dangerous to the health of the consumer, the intake of food without health control. The present study was carried out with the principal objective of identifying and quantifying by Real Time PCR the presence of Salmonella sp. and 16S bacterial rDNA in samples of food adjacent to the Salesian Polytechnic University. About ten establishments were investigated and samples were analyzed in triplicate for subsequent statistical analyzes. As a result, the presence of 16S bacterial rDNA was detected in 100% of the samples using Kruskall-Wallis non-parametric statistical test, which showed statistical significance; while the evaluation of the SPI-1 sequence, a virulence gene for Salmonella sp., 6.7% of positive cases and it was revealed that there is no significant difference. By means of the standard curves of each sequence, the bacterial load for 16S was quantified in an average of 51 ug/mL and in the case of Salmonella sp. A concentration in the range of 0.766 to 0.0170 ug/mL.
Keywords: Salmonella sp., bacterial 16S rDNA, real-time PCR, foods.
_____________________________________________________________________________________________________________________
INTRODUCCIÓN
Las enfermedades transmitidas por alimentos son una gran amenaza a la salud pública en todo el mundo y son una causa importante de mortalidad.1 Aunque muchas son leves y se asocian a síntomas gastrointestinales básicos, en algunas ocasiones las enfermedades transmitidas por agua y alimentos (ETA´s) pueden resultar mucho más peligrosas para la vida de los consumidores en edades tempranas.2
El género Salmonella constituye uno de los grupos de microorganismos patógenos más relevantes en el mundo, provocando serias enfermedades gastroenteríticas en humanos y animales.3 La principal fuente de contagio es la ingesta de comida infectada; algunos productos pueden ser carnes, aves, lácteos, huevos y verduras mal lavadas y preparadas; ya que un incorrecto procesamiento contamina los alimentos.4
Por su parte, ADNr 16S bacteriano es una secuencia ampliamente utilizada como un marcador evolutivo que se encuentra presente en todas las células bacterianas; sirve para rastrear carga bacteriana por ejemplo en alimentos y agua; es un gen constitutivo debido a que se presenta consecutivamente y tiene un tamaño adecuado para realizar análisis, a más de ser fácil de aislar y caracterizar.5
La presente investigación muestra la carga bacteriana y la presencia de Salmonella sp. en muestras de alimentos utilizando la técnica PCR en Tiempo Real como una herramienta sensible, específica y rápida para la determinación de patógenos hasta en mínimas cantidades.
MATERIALES Y MÉTODOS
Localización y tamaño de la muestra
El presente estudio se llevó a cabo en los Laboratorios de Ciencias de la Vida (CIVABI) de la Universidad Politécnica Salesiana Quito, Sede El Girón. Las muestras de alimento fueron tres de diez locales adyacentes a la Universidad.
Recolección de Muestras Para Análisis
“La recolección de las muestras de alimentos se realizó en base a la Norma Técnica Ecuatoriana INEN 1529-2:99 donde se encuentra definida la metodología de muestreo para alimentos de comida rápida”.6
El almacenamiento se lo realizó en conformidad con la norma para continuar con un pre enriquecimiento de las muestras en agua peptonada, lo cual permite conservar las bacterias en óptimas condiciones para los análisis.
Extracción de ADN de las muestras
Utilizando High Pure PCR Template Preparation Kit, se agregó en un tubo eppendorf de 1,5 mL libre de nucleasas, 200µL del material de muestra, para continuar con las indicaciones del fabricante.
Se transfirió la muestra a un tubo de purificación con su filtro específico y se realizó una centrifugación por un minuto a 8000 x g. Para terminar, se agregó 200 µL de buffer de elución precalentado a 70ºC al tubo de filtrado y se almacenó a -20ºC para los análisis posteriores.7
Para la identificación de ADN se utilizó el equipo LightCycler® 2.0 de Roche, con los kits de amplificación de ADN en Tiempo Real.
Técnica PCR en Tiempo Real.
Posterior a la purificación del ADN, se procedió a la aplicación de la técnica de PCR, se colocaron 5 μL de la muestra y se adicionó 10,2 μL de Agua grado Biología Molecular, 0,4 μL del Primer FW, 0,4 μL del Primer RW y 4μL de la Master Mix Sybr Green I (ya adicionada la enzima) en cada capilar con capacidad de 20 μL, se centrifuga a 3.000 rpm durante 5 segundos y se coloca en el carrusel del equipo para realizar la prueba en Tiempo Real.
Los resultados de la amplificación se compararon con un control positivo representado por un estándar bacteriano ATCC, E. coli para determinar ADNr 16S y una cepa certificada de Salmonella para determinar Salmonella sp., cultivados en el laboratorio de Microbiología, finalmente se utilizó un control negativo: 5 μL de agua ultra-estéril.
La técnica de PCR en Tiempo Real se llevó a cabo según el protocolo de LC FS DNA Master PLUS HY-Pb, 96 react. LightCycler (Roche Diagnostics), el cual consiste en 40 ciclos compuestos por cuatro pasos: Desnaturalización: 95ºC, 10 min; Alineamiento: 62ºC, 10sg; Melting: 95ºC, 0; Enfriado: 40ºC, 30sg.
Se realizaron dos Curvas Estándar para cuantificar tanto Salmonella sp. y ADNr 16S utilizando muestras con concentraciones conocidas que fueron previamente cuantificadas en el equipo fluorimétrico: Qubit. Los datos fueron incorporados al software del LightCycler, para así poder cuantificar cada una de las muestras desconocidas.
RESULTADOS Y DISCUSIÓN
Cuantificación mediante PCR en tiempo real de ADNr 16S en las muestras de alimento
De cada una de las muestras de hamburguesa analizadas en esta investigación, se logró detectar mediante la técnica un total de 100% de casos positivos para esta secuencia.
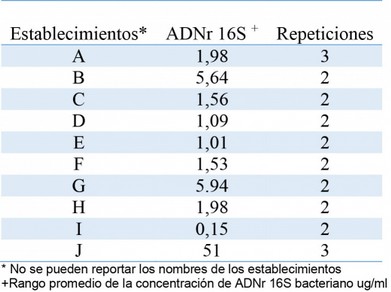
Tabla 1. Casos positivos de ADNr 16S bacteriano en ug/ml por establecimiento
En la Tabla 1 se observa que el establecimiento J presentó la mayor cantidad de concentración de ADNr 16S bacteriano frente a los demás lugares, se cree que esto se debió a que el lugar de expendió se ubicaba en un sitio inapropiado en la vía pública siendo así vulnerable a los patógenos, toxinas o sustancias químicas a más del inadecuado aseo de los manipuladores, la limpieza y la desinfección de insumos y materiales empleados en la preparación de los alimentos, constituyeron factores determinantes que generaron en este análisis un escenario propicio para la contaminación y la propagación bacteriana en el alimento.
Los datos obtenidos de la concentración promedio de la secuencia ADNr 16S fueron analizados mediante el programa estadístico Infostat y SPSS v.17, expresando un resultado positivo, utilizando la prueba estadística de Kruskall-Wallis de análisis de datos no paramétricos, se determinó un H igual a 10,05 lo que determina que si existen diferencias significativas, lo cual permite aceptar la hipótesis alternativa en la que se esperaba que en al menos uno de los establecimientos exista gran carga bacteriana presente en el alimento.8
Según el Instituto Ecuatoriano de Normalización,9 para alimentos preparados, el límite permisible para identificar el nivel aceptable de buena calidad del alimento, debe ser < 3 ufc/g de alimento. Sin embargo, no todos los establecimientos de comida rápida cumplen con esta normativa, como lo menciona Chiluisa-Utreras,6 en donde se realizó un estudió microbiológico y molecular constatando la alta carga microbiana en los alimentos analizados.
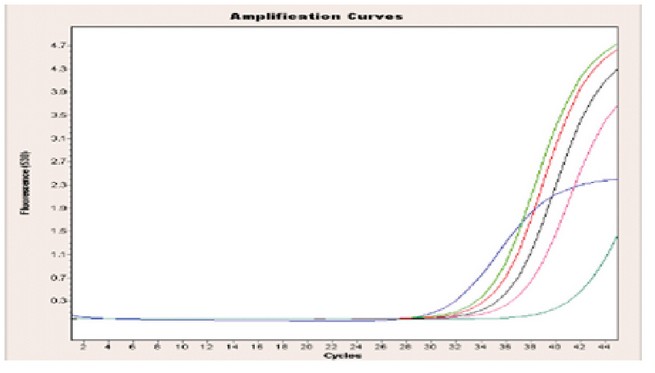
Figura 1. Curvas de amplificación de ADNr 16S bacteriano: fluorescencia vs ciclos
Tras el análisis de las muestras de alimentos, se detectó que existen muestras positivas, como se presenta en la Figura 1, la interpretación de los resultados se llevó a cabo mediante la curva de amplificación, con el punto denominado Cp, éste indica el ciclo en el que la fluorescencia alcanza el valor del umbral presentando picos con fragmentos distintos de ADN que se habían producido en la reacción.
Así, podemos identificar algunas de las curvas obtenidas de cada una de las muestras de alimento para la detección y cuantificación de la secuencia ADNr 16S, en donde la fluorescencia es detectada desde el ciclo 25 con valores altos de concentración, con lo que se afirma que se tiene una carga bacteriana importante debido a que la relación de los ciclos y la carga bacteriana es inversamente proporcional, un Cp bajo presentará concentraciones altas, determinándose de esta manera la presencia y cantidad del patógeno.
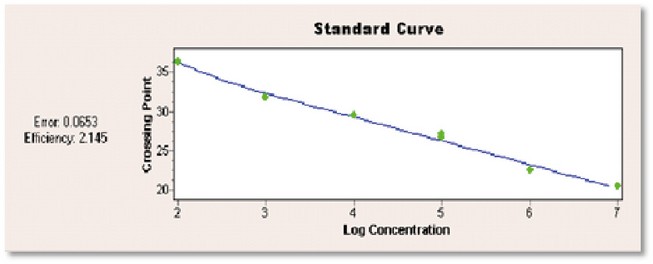
Figura 2. Curva Estándar obtenida para la cuantificación de Salmonella en muestras de alimentos.
Cuantificación mediante PCR en tiempo real de Salmonella sp. en las muestras de alimento
En la Figura 2 se adjunta una de las curvas estándar que se realizó para esta investigación, ésta es generada para la cuantificación de Salmonella en muestras de alimentos; la eficiencia de esta curva es de 2.145 rango que se encuentra dentro de los parámetros aceptables, además presenta un error bajo de 0.0653 puntos es decir cercano a cero, que nos da la certeza en los resultados obtenidos. La cuantificación de ADN de Salmonella sp. por medio de la PCR en tiempo real con la curva de calibración permitió determinar las concentraciones en cada muestra analizada. En la Tabla 2 se muestran los rangos promedios para Salmonella sp. en ug/mL por cada uno de los establecimientos:
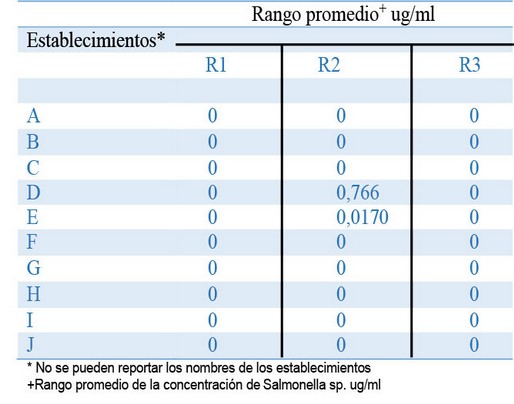
Tabla 2. Concentraciones promedio de Salmonella sp. en ug/ml
Los resultados para las concentraciones de Salmonella que se muestran en la Tabla 2 son cero para la mayoría de los establecimientos analizados, sin embargo una repetición de los establecimientos D y E, presentaron resultados positivos con una concentración de 0,766 y 0,0170 ug/mL; por lo que se logró determinar a través del método un total del 6,7 % de casos positivos, que analizados por los programas estadísticos expresan un resultado negativo utilizando Kruskall-Wallis; se determinó un H igual a 1,56 que determina que no existe diferencia significativa, lo que permite aceptar la hipótesis nula en el que ningún establecimiento presenta carga importante para Salmonella en el alimento.
Salmonella sp. es considerada una bacteria patógena que puede causar enfermedades gastrointestinales,10 su presencia en alimentos debe ser nula, sin embargo, la mala manipulación de alimentos y la poca higiene con la que se los procesa tiene gran incidencia.11
El resultado de una corrida de PCR en tiempo real se visualiza en un gráfico de amplificación. En este caso, los resultados de la amplificación de las corridas de las muestras de alimento son negativas, lo que significa que el valor del umbral no fue superado respecto al control.12
Aplicar esta técnica molecular para la determinación de bacterias causantes de ETA´s, presenta varias ventajas sobre los análisis microbiológicos. Como lo reporta Chiluisa-Utreras,6 la PCR en tiempo real al ser un espectrofluorómetro presenta mayor precisión ya que detecta mínimas cantidades bacterianas, mayor sensibilidad por los fluorocromos que utiliza, especificidad por los primers, y óptima capacidad para procesar grandes cantidades de muestras en poco tiempo ya que el proceso tarda en promedio 24 horas en contraste con el método convencional; además hay que destacar que los métodos moleculares presentan menor cantidad de contaminación que los métodos microbiológicos.
CONCLUSIONES Y RECOMENDACIONES
En conclusión, los resultados obtenidos permiten determinar que la técnica molecular presenta muchas más ventajas para la detección rápida y precisa de patógenos en alimentos y sobre todo para el análisis de la distribución de productos inocuos para los consumidores.13
El desarrollo de métodos moleculares tanto para la detección e identificación de microorganismos patógenos han proporcionado nuevas herramientas confiables para determinar en menor tiempo el riesgo de infección por patógenos en alimentos, por lo que es considerada un instrumento poderoso en el diagnóstico microbiológico.14
Los resultados de esta investigación confirman un alto índice de patógenos en el alimento analizado, la hamburguesa, el cual corresponde a varios establecimientos tanto ambulantes como restaurantes, demostrando que estos lugares no cuentan con una norma de higiene establecida que les permita tener noción de la inocuidad que se necesita para expender un alimento saludable al consumidor.15
Es importante que los organismos de control sanitario realicen inspecciones periódicas a los establecimientos con la finalidad de prevenir enfermedades transmitidas por alimentos contaminados por patógenos, puesto que los mismos están dirigidos a personas jóvenes que buscan alimentos de rápido acceso y consumo.16
Finalmente, se recomienda seguir en la investigación con patógenos alimentarios ya que al obtener un 100% de casos positivos para la secuencia ADNr 16S se analiza que a pesar de no encontrarse mayor carga de Salmonella, estamos al descubrimiento de cualquier otro tipo de bacterias que podrían generar ETA´s.17
AGRADECIMIENTOS
A la MSc. Ivonne Vaca por su aporte en el análisis de datos.
Referencias
1. Ministerio de Salud Pública. (2012). Normas Del Sistema Integrado De Vigilancia Epidemiológica Del Ecuador (Sive). En línea: https://aplicaciones.msp.gob.ec/salud/archivosdigitales/documentosDirecciones/dnn/archivos/NORMA%20sive%208-04-2013.pdf Consulta: 15 de Septiembre 2014.
2. Arenas, L.; Rodas, A.; Narváez, C.; Jerez, N.; Huerta, N.; Giuffrida, M. y Uzcátegui, S. (2009). Calidad microbiológica de las carnes y de los alimentos. AGROTECNICO No. 25. Pág. 44.
3. Dirección Provincial de Salud de Pichincha. (2012). Reporte de los casos de Enfermedades Transmitidas por Agua y Alimentos en la Provincia de Pichincha durante 2011 y 2012. Quito.
4. Jiménez, J., Muñoz, A., & Delgado, D. (2014). Fiebre tifoidea y otras infecciones por Salmonella. México: Facultad de Medicina Universidad Autónoma de México.
5. Clarridge JE. (2004).Impact of 16S rRNA Gene Sequence Analysis for Identification of Bacteria on Clinical Microbiology and Infectious Diseases. Clin Microbiol. doi: 10.1128/CMR.17.4.840-862.2004.
6. Chiluisa-Utreras V., J. Coba y A. Echeverría. (2014). Determinación por PCR en Tiempo Real de Escherichia coli en muestras de comida rápida. La Granja: Revista de Ciencias de la Vida. Vol. 19(1): 44-50. ISSN: 1390-3799.
7. Roche Diagnostics (2010). LightCycler® foodproof Salmonella sp. detection kit. GmbH, Mannheim, Germany.
8. Sánchez-Otero. 2013. Introducción a la estadística en las ciencias biológicas. Quito - Ecuador.
9. Instituto Ecuatoriano de Normalización (INEN) Límite permisibles para alimentos de Salmonella sp. y carga microbiana. En Línea: http://www.normalizacion.gob.ec/ Consulta 12 de Septiembre 2014.
10. Lupien, J., & Flores, M. (2002). Sistemas de Calidad e Inocuidad de los Alimentos. Manual de capacitación sobre higiene de los alimentos y sobre el sistema de Análisis de Peligros y Puntos Críticos de Control (APPCC). Roma: Grupo Editorial, Dirección de Información de la FAO.
11. Rodríguez, R. (2011). Programa Nacional para la Vigilancia, Control y Erradicación de Salmonella en Aves de Ecuador. Quito: Editorial Universitaria.
12. Edwards, K.J. (2004). Performing Real-Time PCR. In: Real-Time PCR; An Essential Guide. K.J.Edwards et al., editors. Horizon Bioscience.Wymondham, p. 71-84.
13. Yánez, E., Máttar, S., & Durango, A. (2008). Determinación de Salmonella sp. por PCR en tiempo real y método convencional en canales de bovinos y en alimentos de la vía pública de Montería, Córdoba. Bogotá: Biblioteca Nacional de Salud “José Celestino Mutis”.
14. Valasek, M. A. and J. J. Repa (2005). "The power of real-time PCR." Adv Physiol Educ29(3): 151-159.
15. Bustin, S.A. (2005). Real-Time PCR. In: Encyclopedia of Diagnostic Genomics and Proteomics M.Podda & J.Fuchs, editors. Marcel Dekker. New York, p. 1131-1135.
16. Ortiz, R.; Gutiérrez, R.; Vega, S.; Díaz, G. y Schettino, B. (2008). Contaminación de los alimentos. Universidad Autónoma Metropolitana Unidad Xochimilco. México. Pág. 7. Publicado en revista virtual RECITELA - v.8 n.1.
17. Chan, O, Wolf, M., Hepperle, D. and Casper, P. (2002). Molecular techniques: An overview of methods for the detection of bacteria. African J. of Biotechnology 2(12):710-713.En línea: http://www.ajol.info/index.php/ajb/article/view/14906/58650.
Recibido: enero 2017
Aprobado: febrero 2017
Viviana Chiluisa-Utreras 1; Andrea Echeverría 1
1Laboratorios de Ciencias de la Vida CIVABI, Universidad Politécnica Salesiana, Quito – Ecuador. Autor para correspondencia: [email protected]
