2020.05.01.13
Files > Volume 5 > Vol 5 No 1 2020
NEWS AND VIEWS /NOTICIAS Y OPINIONES
Los micro ARNs en patología humana: utilidad clínica y enfoque traslacional
The micro RNAs in human pathology: clinical utility and translational approach
Jorge Luis Vélez1, Pablo Morocho2, Mario Montalvo3, Santiago Aguayo4, Pablo Andrés Vélez5, Gustavo Velarde6, Fernando Jara7, César Paz y Miño8.
Available from: http://dx.doi.org/10.21931/RB/2020.05.01.13
RESUMEN
En clínica humana, patologías tan diversas como el cáncer, la sepsis, enfermedades autoinmunes, entre otras; de etiología diferente y un comportamiento fisiopatológico distinto, convergen en un fallo de represión génica que permite la expresión fenotípica de la enfermedad; la posibilidad de tener un marcador biológico que le muestre al clínico estos sucesos es deseable, ya que permitiría tomar estrategias diagnósticas y terapéuticas de forma precoz. Los micro ARNs, son ARNs pequeños y no codificantes que cumplen ese rol “silenciamiento genético”, sin embargo, el paso de la investigación básica a la aplicabilidad clínica, es decir, su utilidad traslacional es aún poco difundido en especialidades diferentes a la Oncología. El objetivo de esta revisión, es explicar de la forma más clara, la utilidad actual de los micro ARNs en diversas enfermedades humanas.
Palabras clave: micro ARNs, enfoque traslacional, clínica humana.
ABSTRACT
In human clinics, pathologies as diverse as cancer, sepsis, autoimmune diseases, among others; of different etiology and a different pathophysiological behavior, converge in a failure of gene repression that allows the phenotypic expression of the disease; The possibility of having a biological marker that shows these events to the clinician is desirable since it would allow early diagnostic and therapeutic strategies. Micro RNAs are small and non-coding RNAs that fulfill that “genetic silencing” role, however, the step from basic research to clinical applicability, that is, their translational utility is still little diffused in specialties other than oncology. The objective of this review is to explain in a more precise way.
Keywords: micro RNAs, translational approach, human clinic.
INTRODUCCIÓN
En las últimas dos décadas, de forma inicial a través de un nemátodo Caenorhabditis elegans (C. elegans) se descubre otro mecanismo poderoso de regulación de la expresión génica a nivel de ARN1, pequeñas moléculas de ARN denominadas micro ARNs (miARN) los cuales han sido sometidos a intensa investigación y su presencia en la clínica humana parece ser universal, desde el cáncer hasta la patología cardíaca, respiratoria, inmune, infecciosa entre otras2,3.
Son ARNs pequeños (19 a 24 nucleótidos), no codificantes y no modificados desde el punto de vista evolutivo y cada miARN tiene el potencial de afectar a 500 genes4; su papel fisiológico fundamental es la de reprimir genes implicados en la proliferación, apoptosis y diferenciación celular5, consecuentemente ‘silenciamiento de la expresión genética’.
Se forman de la siguiente manera (Figura 1):
Empieza en el núcleo celular mediante la acción de la miARN polimerasa II sobre los genes de miARN, de ésta forma mediante transcripción (transeferencia de la información del ADN al ARN), se obtienen pro-miARN6. Sobre el pro-miARN actúa la endonucleasa ARNsa III, conocida como DROSHA y se genera el pre-miARN, para ello DROSHA requiere el cofactor DiGeorge Syndrome Critical Region 8 (DGCR8), todo esto a nivel nuclear6. Formado ya el miARN, sale del núcleo al citoplasma mediante un transportador, la exportina 5, ahí, mediante una segunda endonucleasa ARNsa III (DICER), el mi-ARN se escinde a miARN maduro (doble cadena) miARN (cadena pasajera), la cadena pasajera se degradará y el miARN maduro se someterá a silenciamiento genético mediante ARNs interferentes7. Los miARNs así formados, abandonaran la célula mediante dos formas de liberación: Pasiva; cuando la célula muere de forma programada (cuerpos apoptóticos)y Activa; por secreción celular de exosomas, complejos de ribonucleoproteínas, lipoproteínas de alta densidad y micro vesículas.
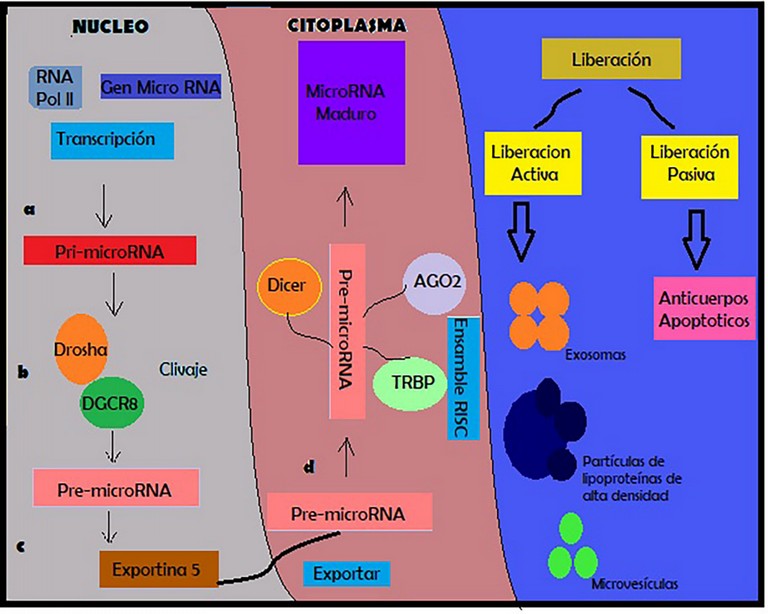
Figura 1. Formación de los mi-ARNs.
Los miARN a su vez interactúan con ARN circulares (ARNcirc), los cuales constituyen moléculas de ARN unidas por un proceso celular denominado splicing o trans-splicing8 y juegan un rol regulatorio importante. Evidencia reciente ha demostrado que los ARNcirc regulan la función de los miARNs9, es por esto que la identificación y estudio de ARNcirc es trascendental para el entendimiento de los mecanismos regulatorios de ciertas enfermedades y para su posterior uso como marcadores biológicos en diagnósticos clínicos.
Asociación ARNcirc-miARN y su importancia reguladora.
Los ARNcirc fueron descubiertos inicialmente en viroides, patógenos de plantas superiores, conformados por una molécula de ARN cerrada covalentemente 8,10. Evidencia reciente demuestra que la expresión de ARNcirc en mamíferos es más prevalente y abundante de lo que se pensaba 11,12, siendo altamente conservados evolutivamente 13; al contrario de los miARNs (moléculas que regulan la expresión de la mayoría de ARN mensajeros 14), los ARNcirc no han sido muy estudiados.
Los ARNcirc juegan un rol regulatorio importante, interactuado con ARN mensajeros (ARNm), microARN o proteínas de unión con ARN (RBPs) 15. (Figura 2).
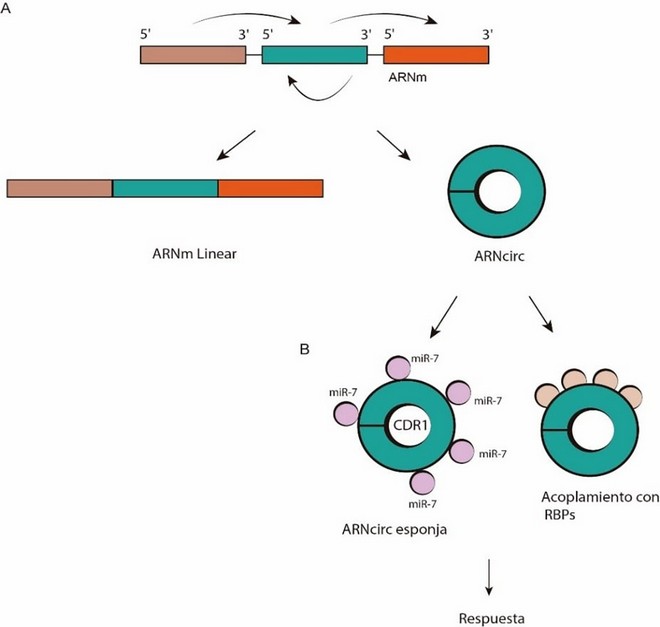
Figura 2: A. Formación de los ARN circulares, B. relación con microARN y acoplamiento con RBPs.
Función reguladora: ARNcirc esponja.
La principal función reguladora de los ARNcirc descubierta hasta el momento es su mecanismo de acción a manera de esponja, secuestrando miRNAs para regular su función 8,16. El CDR1 es quizás el ARNcirc mamífero mejor caracterizado, muy abundante en neuronas 15,16, el ARNcirc CDR1 recientemente identificado como regulador negativo del microARN miR-7, demuestra el potencial regulador (ESPOJA) de estos ARN 8. miR-7 es desestabilizado por un mecanismo que modifica los terminales 5' y 3' del miARN, añadiendo o removiendo nucleótidos 17. (Figura 3).

Figura 3: Función de ARNcirc A. ARNcirc CDR1 actúa como esponja, uniéndose a miR-7 en condiciones normales, previniendo su unión con el ARNm lo que conlleva a su transcripción normal. B. En enfermedad ARNcirc no cumple completamente con su función de esponja liberando miR-7 que se unen al ARNm provocando el silenciamiento de ese gen.
Otro ejemplo de estos ARN circulares como esponja es el HRCR (ARNcirc relacionado al corazón)18. Estudios sugieren que este juega un rol protectivo en hipertrofia cardiaca secuestrando miR-233 19.
Función de acoplamiento para RBPs.
Los ARNcirc también pueden funcionar como sitios de acoplamiento para RBPs para secuestrarlos de su ARN objetivo o para mediar su localización subcelular. Curiosamente, los ARNcirc son particularmente abundantes en el cerebro humano (sinapsis) comparado con otros tejidos analizados 20, sugiriendo su participación en la regulación de ARN locales. Por ejemplo: ARNcirc deben transportar miARNs y RBPs a sitios de sinapsis y liberarlos en respuesta a un especifico estimulo 21.
Aplicabilidad clínica de los Micro ARNs.
El sello distintivo de la función de miARN es su capacidad para suprimir la expresión génica mediante la unión de sus ARN diana, la expresión aberrante de los miARNs afecta la regulación de muchas funciones celulares y redes de genes. Se ha encontrado en tejidos y en sueros de pacientes, como biomarcadores de pronóstico, los miARN también permiten la predicción del curso de una enfermedad22.
Además del cáncer, existe evidencia suficiente para sugerir que los miARNs están desregulados en infecciones virales, trastornos del sistema nervioso, trastornos cardiovasculares, musculares, diabetes y otras enfermedades lo que implica que la utilización de estos miARNs expresados de forma aberrante como biomarcadores para enfermedades es una valiosa estrategia de diagnóstico.
Sepsis.
Los miARNs miR-221 y miR-222 en ratones en quienes se administró lipopolisacárido (un patrón molecular asociado a patógeno PAMP propio de las bacterias gram negativas) mostraron un incremento importante, situación similar se produjo al medirlos en 30 pacientes con sepsis, en ellos, miR-221 y miR-222 se mostraron como reguladores de la reprogramación funcional de macrófagos durante la tolerización (inducción de toleracia) a lipopolisacáridos. La estimulación prolongada con lipopolisacárido condujo a una mayor expresión de miR-221 y mir-222, que regulan el gen 1 relacionado con Brahma (Brg1, también conocido como Smarca4). Este aumento de la expresión provoca el silenciamiento transcripcional de un subconjunto de genes inflamatorios que dependen de la remodelación de la cromatina mediada por SWI / SNF (interruptor / sacarosa no fermentable) y STAT (transductor de señal y activador de la transcripción), que a su vez promueve la tolerancia7,23.
Niveles elevados de éstos marcadores genéticos se correlacionaron con inmunocompromiso y mayor fallo multiorgánico, de ésta forma se demostró que los miARNs miR-221 y miR-222 podrían servir cómo biomarcadores de inmunoparálisis y mal pronóstico en pacientes sépticos7. Tal vez un limitante de los miRNAs citados, es que también marcan el comportamiento biológico de otras patologías como el cáncer, ya que han sido estudiados como biomarcadores en cáncer de mama, cáncer de pulmón de células no pequeñas, glioblastoma, entre otros, lo que significaría poca especificidad7,24.
Caserta et al. evalúa miARNs circulantes relacionados con la inflamación en pacientes con estados inflamatorios infecciosos y no infecciosos, identifica seis principales (miR-30d-5p; miR-30a-5p; miR-192-5p; miR-26a-5p; miR-23a-5p; miR-191-5p), determina que en cualquier estado inflamatorio hay descenso de éstos miARNs, pero que es más importante en sepsis (p: <0,0001) y que el comportamiento biológico hacia la baja de los miARNs discrimina adecuadamente estados sépticos severos de estados inflamatorios graves no infecciosos (AUC: 0,742-0,917). Encuentra además correlación inversa de los niveles de miARNs con los niveles de citoquinas proinflamatorias (IL-1, IL-6, etc). Es decir que la disminución de los miARNs en estados sépticos permite la expresión de citoquinas proinflamatorias, por ausencia de silenciamiento. Entonces a menores niveles de miARNs mayor severidad inflamatoria, con mayor capacidad predictiva en estados inflamatorios secundarios a sepsis23.
Enfermedades cardiovasculares.
Un número cada vez mayor de estudios ha demostrado que los miARNs cardiacos son notablemente alterados en la isquemia miocárdica así como en la remodelación ventricular y la reparación cardíaca. Se encontró que la expresión de miARN-320 estaba sistemáticamente desregulada en los corazones isquémicos4. La reducción del miARN320 endógeno brindó protección contra la apoptosis de los cardiomiocitos a través de la regulación positiva de la HSP2012,22.
Uno de los miARN más estudiados en este ámbito es el miARN-132, el cual en modelos farmacológicos, las reacciones de miARN-132 muestran la eficacia terapéutica para bloquear el desfibrilación cardiaca por la normalización del tamaño cardiaco y la inhibición de la fibrosis25. Los miembros de la familia de miARN-34 promueven la detención del crecimiento y la apoptosis; la inhibición terapéutica de miARN-34 remodelación inducida por isquemia y mejora de la recuperación cardíaca26.
La reperfusión del corazón isquémico sigue siendo la intervención más efectiva para mejorar los resultados clínicos, sin embargo, los aumentos anormales en el calcio intracelular durante la reperfusión miocárdica pueden causar la muerte de cardiomiocitos, conocida como lesión por isquemia reperfusión, el miARN-214 está regulado al alza durante la lesión isquémica y la eliminación genética de miARN-214 en ratones causó una pérdida de contractilidad cardíaca, aumento de la apoptosis y fibrosis excesiva en respuesta a la lesión. Las funciones cardioprotectoras de miARN-214 durante la lesión por isquemia reperfusión se atribuyeron a la represión del ARN mensajero que codifica el intercambiador de sodio / calcio, un regulador clave del flujo de Calcio; y a la represión de varios efectores posteriores de la señalización del calcio que median la muerte celular27. Estos resultados sugirieron un papel fundamental para el miARN-214 como regulador de la homeostasis y la supervivencia de los cardiomiocitos.
Cáncer
La expresión de miARNs en el cáncer se debe a que la mayoría está localizado en regiones genómicas relacionadas con su desarrollo. Estudios recientes también han demostrado sus papeles clave en la evolución patogénica, la progresión y la metástasis de los carcinomas28.
El perfil de expresión de miARN ha sido evaluado recientemente como un biomarcador de diagnóstico confiable para diferenciar entre muestras normales y tumorales. Además de distinguir los tejidos normales de los cancerosos, los marcadores moleculares también se pueden usar para determinar el tejido de origen en tumores de origen primario desconocido29. La expresión de miARN mejora (miARN oncogénico) o reduce (supresor tumoral) el crecimiento tumoral a medida que el tumor progresa y se encontró que está asociado con la resistencia a los medicamentos.
Existen datos que demuestran que su expresión anormal es una característica de los procesos neoplásicos (Tabla 1).
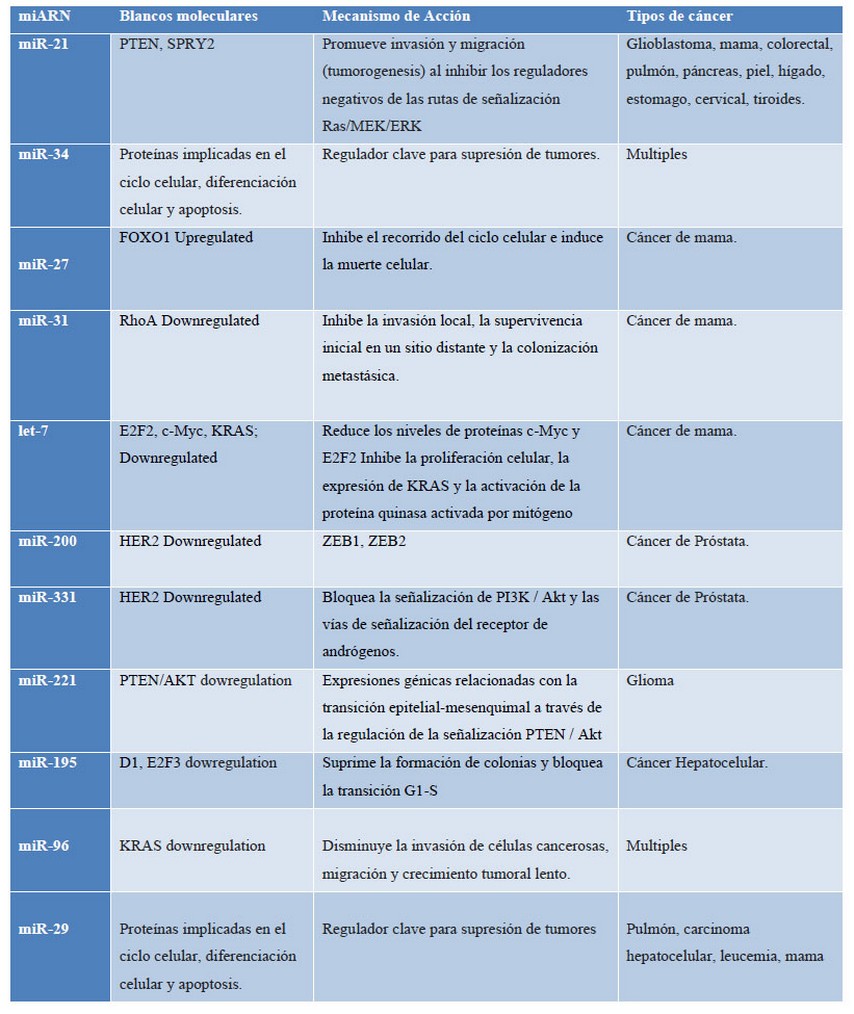
Tabla 1: miARN relacionados con distintos tipos de cáncer.
FOXO1/O3 proteína forkhead; RhoA, Gen homologo familiar A; HER3, receptor de proteína tirosina – quinasa erbB – 3; PTEN, fosfatasa y tensinogeno homologo; AKT, proteína quinasa B, E2F2, transcriptor del factor E2F2.
Diabetes
Los ARN que interactúan con proteínas Piwi (piARN), los pequeños ARN endógenos de interferencia (iARN), los micro-ARN derivados de intrones (miARN) y una serie de ARN no codificantes ejercen una función reguladora, aunque los que mejor se conocen en lo referente a las complicaciones diabéticas son los miARN, que regulan varias vías biológicas clave y funciones celulares implicadas en las complicaciones diabéticas30. Entre ellas se cuentan los elementos reguladores de las concentraciones de especies reactivas del oxígeno (ROS), la conexión subyacente entre la hiperglucemia intracelular y las vías que causan complicaciones derivadas de esta patología.
Estudios demuestran que los miARN intervienes en el desarrollo del páncreas, así como para la regulación de la glucosa y contribuyen en el control de las células β, con una participación activa en la regulación de la producción, secreción y acción de la insulina30,31 (Tabla 2).

Tabla 2: miARN relacionados con la diabetes.
Lupus eritematoso sistémico.
Se ha publicado la expresión diferencial de los micro-ARN en múltiples tipos de células en pacientes con LES. Debido a que la expresión de miARN está involucrada en la regulación de múltiples aspectos de la respuesta inmunitaria normal, no es sorprendente que los cambios en el contexto del miARN puedan asociarse con la autoinmunidad (20).
La sobreexpresión de varios miARN en los linfocitos T CD4 de los pacientes con LES contribuye al defecto de metilación del ADN, en tal razón la expresión de miARN-126 y miARN-148ª, ambos con efecto al inhibir el DNMT1 favoreciendo a la expresión de la enfermedad (21). Otro miARN asociado al LES es miR-146a, que tiene como objetivos directos varios genes relacionados con el IFN, como IRAK1, TRAF6, IRF5 y STAT1 (22). La inhibición de miR-146a provoca una mayor activación del IFN de tipo I, un rasgo de la patogenia característico del LES.
Herramientas bioinformáticas y base de datos disponibles
Actualmente, no existe un enfoque sistemático disponible para identificar ARN circulares en el transcriptoma humano. Debido a que estas moléculas tienen un rol crítico en medicina y en biología, varias bases de datos se han creado para simplificar sus características y funciones.
- CircBase: base de datos que cubre información de ARNcirc, miARN desde el 2013 y frecuentemente se encuentra actualizado con nuevas publicaciones. Se puede acceder a través de http://www.circbase.org/. Esta información se puede descargar y analizar en un contexto genómico, reuniendo datos de varios organismo modelo usados en investigación biomédica.
- StarBase v2.0: esta plataforma explora y predice interacciones ARNcirc-miARN. Se puede acceder a través de http://starbase.sysu.edu.cn/, siendo la primera base de datos que identifica la interacción ARN-ARN y proteína-ARN usando análisis CLIP-seq.
CONCLUSIONES
Los miARNs son ARNs pequeños y no codificantes, su papel fisiológico fundamental es el ‘silenciamiento de la expresión genética’. Se desregulan en varias enfermedades, de diversa etiología, pero de génesis común “falta de represión génica”. Medir estos miARNs expresados de forma aberrante, los hace importantes biomarcadores, que aportan datos diagnósticos y pronósticos valiosos.
Exención de responsabilidad
Los autores confirman que el contenido clínico, diagnóstico y terapéutico del reporte de caso son producto del escrutinio médico aplicado al paciente y son responsabilidad estrictamente de los profesionales involucrados en la publicación de este reporte de caso.
Financiamiento
No hubo financiamiento para la realización de este artículo
Declaración de conflictos de interés
Los autores declaran que no existe conflicto de interés para la publicación de este artículo.
REFERENCIAS
1. Lohr JN, Galimov ER, Gems D. Does senescence promote fitness in Caenorhabditis elegans by causing death? Ageing Res Rev. 2019;50(September 2018):58-71. doi:10.1016/j.arr.2019.01.008.
2. Ullah M, Ng NN, Concepcion W, Thakor AS. Emerging role of stem cell-derived extracellular microRNAs in age-associated human diseases and in different therapies of longevity. Ageing Res Rev. 2020;57:100979. doi:10.1016/j.arr.2019.100979.
3. Ahmed ASI, Sheng MH, Wasnik S, Baylink DJ, Lau K-HW. Effect of aging on stem cells. World J Exp Med. 2017;7(1):1. doi:10.5493/wjem.v7.i1.1.
4. Diab DL, Yerian L, Schauer P, et al. NIH Public Access. 2009;6(11):1249-1254. doi:10.1016/j.cgh.2008.07.016.Cytokeratin.
5. Sen CK. Expanding Horizons of Cellular Plasticity in Regenerative Medicine. Am J Pathol. 2015;185(10):2592-2595. doi:10.1016/j.ajpath.2015.06.003.
6. MT B, K C, D G. Exportin 5 is a RanGTP-dependent dsRNA-binding protein that mediates nuclear export of pre-miRNAs. Rna. 2004;10(2):185. doi:10.1261/rna.5167604.Most.
7. Ceribelli A, Satoh M, Chan EKL. MicroRNAs and autoimmunity. Curr Opin Immunol. 2012;24(6):686-691. doi:10.1016/j.coi.2012.07.011.
8. Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature. 2013;495(7441):333-338. doi:10.1038/nature11928.
9. Rong D, Sun H, Li Z, et al. An emerging function of circRNA-miRNAs-mRNA axis in human diseases. Oncotarget. 2017;5(0):1-11. http://www.oncotarget.com/fulltext/19154.
10. Sanger HL, Klotz G, Riesner D, Gross HJ, Kleinschmidt AK. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures. Proc Natl Acad Sci. 1976;73(11):3852-3856. doi:10.1073/pnas.73.11.3852.
11. Danan M, Schwartz S, Edelheit S, Sorek R. Transcriptome-wide discovery of circular RNAs in Archaea. Nucleic Acids Res. 2012;40(7):3131-3142. doi:10.1093/nar/gkr1009.
12. Vicens Q, Westhof E. Biogenesis of circular RNAs. Cell. 2014;159(1):13-14. doi:10.1016/j.cell.2014.09.005.
13. Pamudurti NR, Bartok O, Jens M, et al. Translation of CircRNAs. Mol Cell. 2017;66(1):9-21.e7. doi:10.1016/j.molcel.2017.02.021.
14. Krek A, Grün D, Poy MN, et al. Combinatorial microRNA target predictions. Nat Genet. 2005;37(5):495-500. doi:10.1038/ng1536.
15. Chekulaeva M, Rajewsky N. Roles of Long Noncoding RNAs and Circular RNAs in Translation. Cold Spring Harb Perspect Biol. 2018:a032680. doi:10.1101/cshperspect.a032680.
16. Hansen TB, Jensen TI, Clausen BH, et al. Natural RNA circles function as efficient microRNA sponges. Nature. 2013;495(7441):384-388. doi:10.1038/nature11993.
17. Duchaine TF, Fabian MR. Mechanistic Insights into MicroRNA-Mediated Gene Silencing. Cold Spring Harb Perspect Biol. 2018:a032771. doi:10.1101/cshperspect.a032771.
18. Lee ECS, Elhassan SAM, Lim GPL, et al. The roles of circular RNAs in human development and diseases. Biomed Pharmacother. 2019;111(December 2018):198-208. doi:10.1016/j.biopha.2018.12.052.
19. Wang K, Long B, Liu F, et al. A circular RNA protects the heart from pathological hypertrophy and heart failure by targeting miR-223. Eur Heart J. 2016;37(33):2602a-2611a. doi:10.1093/eurheartj/ehv713.
20. Rybak-Wolf A, Stottmeister C, Glažar P, et al. Circular RNAs in the Mammalian Brain Are Highly Abundant, Conserved, and Dynamically Expressed. Mol Cell. 2014;58(5):870-885. doi:10.1016/j.molcel.2015.03.027.
21. Ebbesen KK, Kjems J, Hansen TB. Circular RNAs: Identification, biogenesis and function. Biochim Biophys Acta - Gene Regul Mech. 2016;1859(1):163-168. doi:10.1016/j.bbagrm.2015.07.007.
22. Wang J, Chen J, Sen S. MicroRNA as Biomarkers and Diagnostics. J Cell Physiol. 2016;231(1):25-30. doi:10.1002/jcp.25056.
23. Søndergaard ES, Alamili M, Coskun M, Gögenur I. MicroRNA’s are novel biomarkers in sepsis - A systematic review. Trends Anaesth Crit Care. 2015;5(5):151-156. doi:10.1016/j.tacc.2015.08.001.
24. Sarkar FH, Li Y, Wang Z, Kong D, Ali S. Implication of microRNAs in drug resistance for designing novel cancer therapy. Drug Resist Updat. 2010;13(3):57-66. doi:10.1016/j.drup.2010.02.001.
25. Beermann J, Piccoli M-T, Viereck J, Thum T. Non-coding RNAs in Development and Disease: Background, Mechanisms, and Therapeutic Approaches. Physiol Rev. 2016;96(4):1297-1325. doi:10.1152/physrev.00041.2015.
26. Boon RA, Iekushi K, Lechner S, et al. MicroRNA-34a regulates cardiac ageing and function. Nature. 2013;495(7439):107-110. doi:10.1038/nature11919.
27. Aurora AB, Mahmoud AI, Luo X, et al. MicroRNA-214 protect. 2012;122(4). doi:10.1172/jci59327ds1.
28. Sikic D, Wirtz RM, Wach S, et al. Androgen Receptor mRNA Expression in Urothelial Carcinoma of the Bladder: A Retrospective Analysis of Two Independent Cohorts. Transl Oncol. 2019;12(4):661-668. doi:10.1016/j.tranon.2019.01.005.
29. Zhu Y, Li T, Chen G, et al. Identification of a serum microRNA expression signature for detection of lung cancer, involving miR-23b, miR-221, miR-148b and miR-423-3p. Lung Cancer. 2017;114(April 2017):6-11. doi:10.1016/j.lungcan.2017.10.002.
30. Singh K, Pal D, Sinha M, et al. Epigenetic Modification of MicroRNA-200b Contributes to Diabetic Vasculopathy. Mol Ther. 2017;25(12):2689-2704. doi:10.1016/j.ymthe.2017.09.009.
31. Guadalupe Rico-Rosillo M, Vega-Robledo GB, Oliva-Rico D, Obesidad N, De Investigación D. Temas de actualidad Importancia de los microARN en el diagnóstico y desarrollo de enfermedades. Rev Med Inst Mex Seguro Soc. 2014;52(3):302-307. http://www.medigraphic.com/pdfs/imss/im-2014/im143n.pdf.
Recibido: 10 diciembre 2019
Aceptado: 20 enero 2020
Jorge Luis Vélez1, Pablo Morocho2, Mario Montalvo3, Santiago Aguayo4, Pablo Andrés Vélez5, Gustavo Velarde6, Fernando Jara7, César Paz y Miño8.
1. Universidad Central del Ecuador-Hospital Pablo Arturo Suárez. (ORCID https://orcid.org/0000-0002-6956-4475) mail: [email protected]
2. Pontificia Universidad Católica del Ecuador. (ORCID: https://orcid.org/0000-0002-7458-4833) mail: [email protected]
3. Hospital Pablo Arturo Suárez. (ORCID: https://orcid.org/0000-0003-2987-7095), mail: [email protected]
4. Hospital Pablo Arturo Suárez. (ORCID: https://orcid.org/0000-0003-4919-5497), mail: [email protected]
5. Universidad Central del Ecuador. (ORCID: https://orcid.org/0000-0002-6392-3895), mail: [email protected]
6. Pontificia Universidad Católica del Ecuador. (ORCID: https://orcid.org/0000-0002-6530-1280), mail: [email protected]
7. Hospital Pablo Arturo Suárez. (ORCID: https://orcid.org/0000-0003-2132-7187), mail: [email protected]
8. Centro de Investigación Genética y Genómica UTE. (ORCID: https://orcid.org/0000-0002-6693-7344). mail: [email protected]
Autor de Correspondencia:
Dr. Jorge Luis Vélez Paez
Facultad de Ciencias Médicas – Universidad Central del Ecuador
Iquique N14-121 y Sodiro – Itchimbía, El Dorado.
Teléfonos: (593-02) 2528 690
Correo electrónico: [email protected]
