2018.03.02.6
Files > Volume 3 > Vol 3 No 2 2018
INVESTIGATION / RESEARCH
Análisis de comunidades bacterianas asociadas a la rizósfera de especies vegetales Miconia zamorensis y Erato polymnoides en suelos contaminados por metales pesados.
Bacterial community analysis associated to rhizosphere plant species of Miconia zamorensis and Erato polymnoides in heavy metals polluted soils.
Manuel Alexander Rojas 1. Oscar Amable Vivanco Galván 2.
Available from: http://dx.doi.org/10.21931/RB/2018.03.02.6
ABSTRACT
Polluted ecosystems has severaL microorganisms such as bacteria can improve biomass production and tolerance to heavy metals and stress condition . In this context, the present study is focus in the identification and comparison of the bacterial communities associated to vegetal species (i.e. Miconia zamorensis and Erato polymnoides). The bacterial isolates were obtained from rhizosphere of polluted heavy metal soils. Molecular analysis was based on the amplification of a partial region of 16S rRNA. The phylogenetic analysis reveal that the isolated microorganisms belong to three genera: Bacillus, Lysinibacillus and Serratia, members of PGPR (plant growth promoting rhizobacteria) bacteria. This information contribute to the knowledge bacterial fraction cultivable, in the future the information can be used in bioremediation projects.
KeyWords: Molecular characterization, 16S rRNA, bioremediation, rhizobacteria, PGPR.
RESUMEN
En ciertos ecosistemas contaminados, varios microorganismos como las bacterias pueden mejorar la producción de biomasa y la tolerancia de las plantas frente a metales pesados como condiciones de estrés. En este contexto el presente estudio busca la identificación y comparación de las comunidades bacterianas que se encuentran asociadas a especies vegetales (i.e. Miconia zamorensis y Erato polymnoides) mediante el empleo de las técnicas moleculares: PCR y secuenciación de Sanger. Las bacterias se obtuvieron a partir de aislamientos a partir de muestras de suelo de la rizósfera contaminados por metales pesados. El análisis molecular se basó en la amplificación de una región parcial del ARNr 16S. El análisis filogenético evidenció que los microorganismos aislados pertenecen a tres géneros: Bacillus, Lysinibacillus y Serratia los cuales son géneros de bacterianas con capacidad PGPR (plant growth promoting rhizobacteria). Con esta información se contribuye al conocimiento de la diversidad cultivable de microorganismos, así mismo a futuro puede ser usada en proyectos para biorremediar suelos contaminados.
Palabras Claves. Herramientas moleculares, 16S ARN ribosomal, , rizobacterias PGPR.
INTRODUCCIÓN
En Ecuador la actividad minera artesanal de pequeña escala genera un alto grado de contaminación ambiental, sobre todo aquella relacionada con la extracción de oro, ya que la extracción de este mineral requiere del uso de metales pesados como mercurio, plomo y sustancias químicas como cianuro, ácido sulfúrico, entre otros (1), los mismos que causan contaminación de todos los ecosistemas contiguos (2).
Ademas, como consecuencia de la actividad humana, se generan concentraciones elevadas de metales pesados residuales en el suelo (3), los cuales son difícilmente biodegradados y por lo tanto se acumulan en el medio ambiente (4), causando cambios en la composición, biomasa y actividad de las comunidades microbianas, lo que resulta en efectos adversos sobre la salud de las plantas, limitando el crecimiento vegetal, la cobertura vegetal, y la calidad de los cultivos (5). También ejercen un efecto perjudicial en la cadena alimenticia de varias especies y causan la muerte de muchos animales que consumen plantas con un alto contenido de metales pesados (3). Adicionalmente, la contaminación de las cadenas alimenticias y las fuentes de agua, tiene efectos adversos sobre la salud humana, ya que muchos de estos metales pesados y metaloides son tóxicos y pueden causar efectos no deseables y problemas severos, incluso a bajas concentraciones (4).
Los desechos de la minería se han convertido en una grave problema en todo el mundo que incrementa cada vez más con la industrialización y que requiere atención inmediata (4). Actualmente las nuevas tecnologías como la biorremediación, apuntan al uso de especies vegetales y microorganismos rizosféricos para remover, contener o retener contaminantes, convirtiéndose en una alternativa de bajo costo y con enormes ventajas ambientales, haciéndola de gran interés en todo el mundo para recuperar suelos contaminados por metales pesados (4,6).
La fitorremediación es una nueva tecnología promisoria que implica el uso de plantas que tienen la capacidad para mejorar el suelo y/o la calidad del agua mediante la inactivación o la translocación de los contaminantes en los diferentes órganos de la planta sin efectos negativos en la actividad biológica, estructura y fertilidad del suelo(7). Sin embargo, el éxito de la fitorremediación también depende de las asociaciones beneficiosas entre los microorganismos del suelo (como las bacterias de la rizósfera) y las plantas, en donde la planta proporciona una fuente de carbono para las bacterias, y a su vez, las bacterias son capaces de producir ciertas substancias o metabolitos, que promueven el crecimiento de la planta, la cual ayuda a disminuir el impacto negativo de la toxicidad por metales pesados (7). Las bacterias también pueden actuar sobre estos contaminantes, a través de la bioacumulación, inmovilización o removiéndolos más fácilmente del ambiente a través de la quelación de metales y la subsecuente eliminación mediante la secreción de exopolisacáridos (8,9).
En base a toda esta problemática, el presente proyecto busca la identificación y comparación de las comunidades bacterianas asociadas a la rizósfera de especies vegetales Miconia zamorensis y Erato polymnoides.
Con esta información, a futuro, se podrá trabajar en aplicaciones biotecnológicas que incluyan el uso de consorcios microbianos con capacidad de biorremediar zonas contaminadas.
MATERIALES Y MÉTODOS
Obtención de aislados bacterianos.
Para el desarrollo del presente proyecto de investigación, la colección de bacterias de la Universidad Técnica Particular de Loja (UTPL) proporcionó aislados bacterianos para la identificación molecular correspondientes a dos áreas de muestreo: zona uno corresponde al vertedero de desechos mineros (contaminado) se eligieron cepas obtenidas de la rizosfera de Miconia y Erato (24 cepas por especie vegetal); la zona dos (control) es un sitio ubicado a 50 metros, de donde se emplearon cepas bacterianas de Miconia y Erato (24 cepas por especie vegetal);. El estudio contempla el análisis de 96 cepas en total.
Reactivación en medio de cultivo líquido.
Las cepas bacterianas proporpocionadas por la colección UTPL fueron reactivadas después de un periodo de tiempo en congelación de 8 semanas, para lo cual, se procedió a inocular 300 µL de cada cultivo bacteriano en un tubo con 10 ml del medio líquido, BBL™ Trypticase™ Soy Broth (TSB), los tubos fueron sellados independientemente con parafilm y cubiertos con papel aluminio para incubarse a 27°C en agitación continua (75 rpm - 24 horas). Esto con el objetivo de incrementar la biomasa bacteriana para la extracción de ADN.
Caracterización molecular de bacterianas.
Se procedió a realizar la extracción de ADN de las 96 cepas bacterianas reactivadas en el medio de cultivo líquido por 24 horas, para ello se empleó el kit de extracción UltraClean Microbial DNA Isolation kit (MoBio) siguiendo el protocolo del fabricante.
Para la identificación de las cepas bacterianas, la región parcial V3 del ARNr 16S fue amplificada mediante PCR aplicando los primers Eub338 5’ -ACT CCT ACG GGA GGC AGC AG- 3’ (forward) y Eub518 5’ -ATT ACC GCG GCT GCT GG- 3’ (reverse) (11) y con la ayuda de un termociclador (Applied Biosystem). Las condiciones de PCR fueron: desnaturalización inicial de 9 min a 94ºC seguido de 30 ciclos de 45 segundos a 94°C, 45 segundos de alineamiento a 55°C, y una extensión de 45 segundos a 72°C, y una etapa de polimerización final de 72°C durante 7 min. Cada reacción (25 µL) contenía: 5 U de GoTag DNA polimerasa (PROMEGA), 5 µL de buffer de reacción 5X, MgCl2 1 mM, 1 µL de ADN molde, 0,2 mM de dNTPs (PROMEGA) y 0,25 µL de cada primer.
Los amplicones de la PCR fueron separados mediante una corrida electroforética a 128 voltios y 300 mA durante 25 minutos en gel de agarosa al 1%, preparado con buffer SB1X y SYBR®Safe para la visualización del ADN en el gel. Las muestras se prepararon tomando 2 µL del producto de la PCR mezclado con 1 µL de buffer (5X Green GoTaq®Flexi Buffer) y se cargaron en cada pocillo del gel junto con 2 µL del marcador de peso molecular 1Kb DNA Ladder (Invitrogen) en el último pocillo.
Una vez terminada la comprobación de todos los productos de la PCR mediante electroforesis, se procedió a realizar la purificación de ADN, para lo cual, se utilizó el kit Wizard® SV Gel and PCR Clean-Up System, siguiendo el protocolo del fabricante.
Los productos de PCR purificados, fueron colocados en una placa de 96 pocillos con un volumen de 15 µL por muestra, se sellaron y se enviaron a la compañía Macrogen, Korea (http://www.macrogen.com/kor/) para la obtención de las secuencias.
Análisis Bioinformático y filogenético.
El análisis fue realizado a partir de las 96 secuencias obtenidas, correspondientes a Erato control (24), Miconia control (24), Erato contaminado (24), Miconia contaminado (24).
Las secuencias obtenidas fueron editadas con el programa BioEdit (Versión 7.2.5) y luego comparadas con secuencias en la base de datos del GenBank utilizando la opción Blast del sitio web NCBI (http://blast.ncbi.nlm.nih.gov/Blast.cgi) para encontrar secuencias estrechamente relacionadas e identificar las cepas bacterianas.
Sobre la base de máxima identidad, fueron seleccionadas las primeras diez secuencias y utilizando la herramienta MAFFT (Versión 7, estrategia G-INS-i), fueron alineadas junto con las secuencias obtenidas de los aislados bacterianos.
El análisis filogenético, se realizó utilizando el software MEGA7. El árbol filogenético fue inferido usando el método de Máxima Verosimilitud. Las divergencias de las secuencias se cuantificaron utilizando el modelo de distancia “Kimura-2-Parameter”. Para el tratamiento de los gaps, se eligió la opción “Use all sites”. Se calcularon un total de 1000 repeticiones de bootstrap.
RESULTADOS Y DISCUSIÓN
Viabilidad de cepas.
Se determinó una viabilidad del 100 % de las cepas usadas luego de ser reactivadas en medios específicos, previo a la extracción de ADN.
Verificación de los amplicones a través de electroforesis.
Un total de 96 muestras fueron analizadas mediante electroforesis y se confirmó que todos los fragmentos de ADN amplificados tienen los tamaños esperados de aproximadamente 200 pares de bases en relación al marcador de peso molecular. La figura 1 muestra los resultados de los productos amplificados por PCR de un grupo de muestras con los primers Eub338 y Eub518.
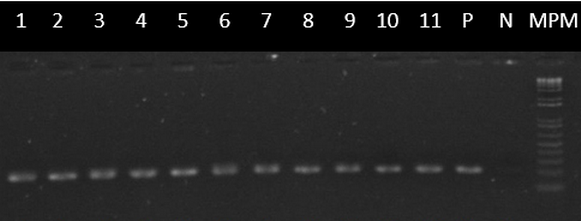
Figura 1: Amplificación por PCR de un fragmento corto de la región 16S en bacterias: 1-2: Muestras de Miconia zamorensis contaminado (MP); 3-5: Muestras de Miconia zamorensis control (MC); 6-8: Muestras de Erato polymnoides contaminado (EP); 9-11: Muestras de Erato polymnoides control (EC); P control positivo; N control negativo; MPM marcador de peso molecular.
Para el desarrollo del presente estudio, se realizó la amplificación de una región parcial del ARNr 16S correspondiente a la región hipervariable V3 del gen, cuyo tamaño aproximado es de 200 pb. Dicha región ha demostrado ser la más destacada para estudios de diversidad bacteriana debido a su mayor variabilidad y secuencia hipervariable más larga (12).
Análisis de secuencias y comparación en base de datos.
Luego de realizar el análisis de cada cromatograma, se determinó que 61 secuencias presentan cromatogramas de buena calidad para el análisis bioinformático. Las 35 secuencias faltantes no se utilizaron para la identificación molecular, ya que estas secuencias presentaban cromatogramas fallidos o irregulares.
Las 61 secuencias de las cepas bacterianas fueron analizadas a través de BLAST, mostrando una similitud del 99 - 100% con las secuencias homologas cercanas a la base de datos del GenBank. La identificación filogenética de los aislados bacterianos, basada en la secuenciación de la región parcial V3 del gen 16S ARNr se muestra en laTabla 1.
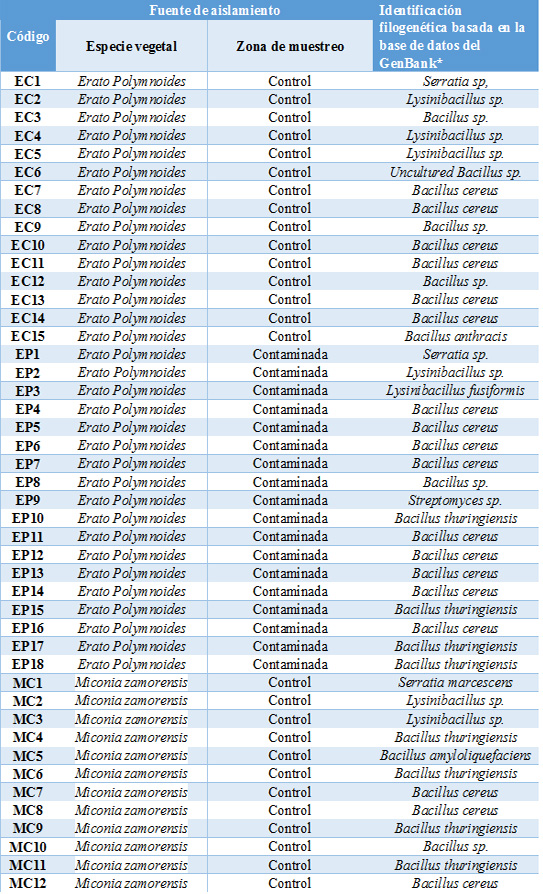
Tabla 1: Comparación de secuencias de aislados bacterianos con BLAST.
Análisis filogenético de Bacterias.
El análisis filogenético basado en la secuenciación del gen ARNr 16S, reveló la similitud de los aislados bacterianos a los géneros Bacillus y Lysinibacillus pertenecientes al filo Firmicutes y Proteobacteria (género Serratia). El árbol construido por el método de Máxima Verosimilitud confirmó la posición filogenética de las cepas bacterianas (Figura 2).

Figura 2: Relaciones filogenéticas basadas en la secuenciación del gen ARNr 16S entre las cepas aisladas y las secuencias de varias especies de referencia recuperadas de la base de datos GenBank del NCBI. El árbol fue generado mediante el método de Máxima Verosimilitud con 1000 réplicas de bootstrap. Solo se muestran los valores de bootstrap ≥ 70%. Los números de acceso al GenBank para las secuencias de referencia se dan después del nombre de la cepa. La especie Alicyclobacillus aeris se utilizó como grupo externo.
En el presente estudio, se ha podido conocer la presencia de los géneros bacterianos Bacillus, lysinibacillus y Serratia, de más frecuente a menos frecuente respectivamente, tanto en la zona contaminada como en la zona control no contaminada (Figura 3 y Figura 4). Estos resultados son comparables con estudios realizados por (13,14) donde mencionan que la abundancia del phylum Firmicutes (géneros Bacillus y Lysinibacillus), se debe a que en el suelo existen condiciones que pueden favorecer el crecimiento de estos microorganismos, tales como, una mayor disponibilidad de nutrientes (carbono orgánico y porcentaje de nitrógeno y fosforo), altos niveles de humedad y salinidad extrema. Además, especies del género Bacillus, son cosmopolitas y se caracterizan por la formación de endosporas que les permite sobrevivir a diversas condiciones inhóspitas por largos periodos de tiempo, razón por la cual pueden ser hallados en la mayor parte de las muestras de suelo y agua en todo el planeta, además de poder prevalecer en el aire y en diversos huéspedes como las plantas (15), en las que también es común encontrar una abundancia de este género en especies vegetales de la división Magnoliopsida (16,17).
La exposición a concentraciones elevadas de metales pesados, puede ser un factor adicional que puede llevar a la selección de estos microrganismos y favorecer a los más tolerantes o resistentes en los suelos de la rizósfera, debido a la rápida adaptación de estos microorganismos a los cambios ambientales (18,19). Varios estudios han reportado que muchas de las especies bacterianas aisladas en este estudio, pertenecientes a los géneros Bacillus, Lysinibacillus y Serratia, están relacionadas con cepas degradantes y resistentes a diferentes metales pesados, entre los cuales se mencionan, Pb, Cu, Ni, Cr, Zn, Cd, Co y As, por lo que tienen un enorme potencial en la biorremediación de la acumulación de metales pesados en el suelo y agua (20–22,17,23–26). Bajo estas condiciones, se ha propuesto que la dominancia de algunos grupos bacterianos se debe a interacciones competitivas en las que algunas especies bacterianas son eliminadas por exclusión competitiva y las especies mejor adaptadas son las que dominan (13).
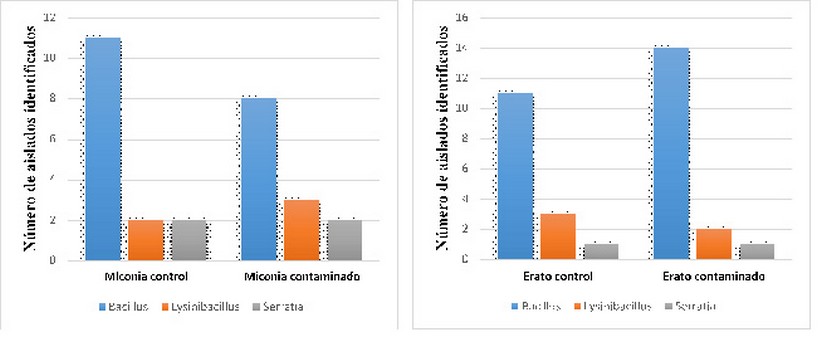
Figura 3 y figura 4: las presentes gráficas, muestran la diversidad y el número total de aislados bacterianos identificados, asociados a la rizósfera de Miconia zamorensis y Erato polymnoides en la zona contaminada y control. El género más predominante en ambos casos fue el género Bacillus, seguido de Lysinibacillus y Serratia.
Como se muestra en las figuras 3 y 4, no se evidencia una mayor diferencia en la distribución de los microrganismos identificados entre ambas áreas de estudio de donde se obtuvieron las cepas bacterianas. Esto puede deberse a que, según menciona (10), tanto el área contaminada como el área control, se encuentran en una zona con alto grado de contaminación ambiental. En el área de muestreo contaminada por metales pesados, se registra una intensa actividad minera relacionada a la extracción de oro a pequeña escala; mientras que el área utilizada como control, pertenece a una zona minera abandonada, en la que las actividades mineras se detuvieron cinco años atrás del presente estudio, es decir que existieron condiciones similares de contaminación ya que según (3), los compuestos inorgánicos, como es el caso de los metales pesados, pueden permanecer por largo tiempo en el suelo debido a que son difícilmente degradables.
Por otra parte, basándonos en la revisión bibliográfica, es interesante mencionar que los géneros Bacillus y Serratia están relacionadas con especies o grupos de bacterias PGPR (por sus siglas en inglés, plant growth promoting rhizobacteria) que tienen la capacidad de producir ciertas sustancias o metabolitos que promueven el crecimiento de las plantas, tales como, el ácido indol acético (IAA), sideróforos y la enzima ácido 1-aminociclopropano-1-carboxílico (ACC), que mejoran el crecimiento, disminuyen el impacto negativo de las concentraciones de metales pesados tóxicos y por lo tanto, aumentan la eficacia de la fitorremediación (16,17,19,22,25,27,28).
En la actualidad, hay una gran atención enfocada en el tratamiento microbiano de los sitios contaminados por metales pesados y existe la necesidad de un mayor conocimiento de cepas bacterianas resistentes a estos metales (7), ya que, las rizobacterias se utilizan en todo el mundo como bioinoculantes para promover el crecimiento y el desarrollo de las plantas bajo diverso estrés como los metales pesados (22).
CONCLUSIONES
El presente estudio permitió identificar una amplia variedad de comunidades bacterianas en el suelo de la rizósfera de las especies vegetales Miconia zamorensis y Erato polymnoides. Esta contrbución puede motivar a los investigadores a unir esfuerzos para usar microorganismos como potenciales elementos en proyectos de biorremediación para descontaminar suelos con metales pesados.
Este reporte sobre la identificación de especies bacterianas ha contribuido con información relevante de las especies dominantes en suelos contaminados, dando el inicio para en una siguiente fase realizar ensayos in vitrocon el objetivo de determinar la capacidad PGPR y de resistencia a metales pesados de estos componentes bacterianas y posteriormente su aplicación en proyectos biotecnológicos para biorremediar suelos contaminados.
AGRADECIMIENTOS
Quiero agradecer de manera muy especial al Bq. Oscar Amable Vivanco Galván por su ayuda y apoyo incondicional durante todo el trascurso de mi trabajo, por su asesoramiento y valiosos consejos en todo el proceso de esta investigación y también al PhD. Aminael Sánchez Rodríguez por la predisposición, sugerencias y apoyo para este trabajo.
REFERENCIAS
1. Conant J, Fadem P. Guía comunitaria para la Salud Ambiental. Berkeley: Hesperian; 2011. 635 p.
2. Buckley DH, Schmidt TM. Diversity and dynamics of microbial communities in soils from agro-ecosystems. Environ Microbiol. 2003;5(6):441 – 452.
3. Durán P. Transferencia de metales de suelo a planta en áreas mineras: Ejemplos de los Andes peruanos y de la Cordillera Prelitoral Catalana [Internet]. Universidad de Barcelona; 2010. Available from: http://diposit.ub.edu/dspace/handle/2445/36122
4. Ali H, Khan E, Sajad MA. Phytoremediation of heavy metals-Concepts and applications. Chemosphere [Internet]. Elsevier Ltd; 2013;91(7):869–81. Available from: http://dx.doi.org/10.1016/j.chemosphere.2013.01.075
5. Guo Z, Megharaj M, Beer M, Ming H, Rahman MM, Wu W, et al. Heavy metal impact on bacterial biomass based on DNA analyses and uptake by wild plants in the abandoned copper mine soils. Bioresour Technol [Internet]. Elsevier Ltd; 2009;100(17):3831–6. Available from: http://dx.doi.org/10.1016/j.biortech.2009.02.043
6. Gonzalez-Chavez M del CÁ. Recuperación de suelos contaminados con metales pesados utilizando plantas y microorganismos rizosféricos. Terra Latinoam. 2005;23(1):29–37.
7. Vigliotta G, Matrella S, Cicatelli A, Guarino F, Castiglione S. Effects of heavy metals and chelants on phytoremediation capacity and on rhizobacterial communities of maize. J Environ Manage [Internet]. Elsevier Ltd; 2016;179:93–102. Available from: http://linkinghub.elsevier.com/retrieve/pii/S0301479716302262
8. Das S, Dash HR. Microbial bioremediation: A potential tool for restoration of contaminated areas [Internet]. Microbial Biodegradation and Bioremediation. Rourkela, Odisha: Elsevier Inc.; 2014. 1-20 p. Available from: http://dx.doi.org/10.1016/B978-0-12-800021-2.00001-7
9. Karigar CS, Rao SS. Role of microbial enzymes in the bioremediation of pollutants : A review. Enzyme Res. 2011;1–11.
10. Ochoa JF, Sánchez-Rodríguez A. Caracterización molecular y criopreservación de hongos y bacterias asociados a la rizósfera de especies vegetales acumuladoras de metales pesados en suelos contaminados por minería. Bionatura [Internet]. 2017;2(2):288–95. Available from: http://revistabionatura.com/2017.02.02.3.html
11. Fierer N, Jackson JA, Vilgalys R, Jackson RB. Assessment of soil microbial community structure by use of taxon-specific quantitative PCR assays. Appl Environ Microbiol [Internet]. 2005;71(7):4117–20. Available from: http://aem.asm.org/content/71/7/4117.short
12. Vasileiadis S, Puglisi E, Arena M, Cappa F, Cocconcelli PS, Trevisan M. Soil bacterial diversity screening using single 16S rRNA gene V regions coupled with multi-million read generating sequencing technologies. PLoS One. 2012;7(8):1–11.
13. Aislabie JM, Jordan J, Barker GM. Relation between soil classification and bacterial diversity in soils of the Ross Sea region, Antarctica. Geoderma. 2008;144(1–2):9–20.
14. Aislabie J, Jordan S, Ayton J, Klassen JL, Barker GM, Turner S. Bacterial diversity associated with ornithogenic soil of the Ross Sea region, Antarctica. Can J Microbiol. 2009;55:21–36.
15. Nicholson WL, Munakata N, Horneck G, Melosh HJ, Setlow P. Resistance of Bacillus endospores to extreme terrestrial and extraterrestrial environments. Microbiol Mol Biol Rev [Internet]. 2000;64(3):548–72. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=99004&tool=pmcentrez&rendertype=abstract
16. Khan M, Sessitsch A, Harris M, Fatima K, Imran A, Arslan M, et al. Cr-resistant rhizo- and endophytic bacteria associated with Prosopis juliflora and their potential as phytoremediation enhancing agents in metal-degraded soils. Front Plant Sci. 2015;5(January):1–10.
17. Luo S, Chen L, Chen J, Xiao X, Xu T, Wan Y, et al. Analysis and characterization of cultivable heavy metal-resistant bacterial endophytes isolated from Cd-hyperaccumulator Solanum nigrum L . and their potential use for phytoremediation. Chemosphere [Internet]. Elsevier Ltd; 2011;85(7):1130–8. Available from: http://dx.doi.org/10.1016/j.chemosphere.2011.07.053
18. Chen M, Xu P, Zeng G, Yang C, Huang D, Zhang J. Bioremediation of soils contaminated with polycyclic aromatic hydrocarbons, petroleum, pesticides, chlorophenols and heavy metals by composting: Applications, microbes and future research needs. Biotechnol Adv [Internet]. Elsevier Inc.; 2015;33(6):745–55. Available from: http://dx.doi.org/10.1016/j.biotechadv.2015.05.003
19. Zhang W, Huang Z, He L, Sheng X. Assessment of bacterial communities and characterization of lead-resistant bacteria in the rhizosphere soils of metal-tolerant Chenopodium ambrosioides grown on lead – zinc mine tailings. Chemosphere [Internet]. Elsevier Ltd; 2012;87(10):1171–8. Available from: http://dx.doi.org/10.1016/j.chemosphere.2012.02.036
20. Tomova I, Stoilova-Disheva M, Vasileva-Tonkova E. Characterization of heavy metals resistant heterotrophic bacteria from soils in the Windmill Islands region, Wilkes Land, East Antarctica. Polish Polar Res. 2014;35(4):593–607.
21. Gupta K, Chatterjee C, Gupta B. Isolation and characterization of heavy metal tolerant Gram-positive bacteria with bioremedial properties from municipal waste rich soil of Kestopur canal (Kolkata), West Bengal, India. Versita. 2012;67(5):827–36.
22. Kumar V, Singh S, Singh J, Upadhyay N. Potential of plant growth promoting traits by bacteria isolated from heavy metal contaminated soils. Bull Environ Contam Toxicol. 2015;
23. Pepi M, Volterrani M, Renzi M, Marvasi M, Gasperini S, Franchi E, et al. Arsenic-resistant bacteria isolated from contaminated sediments of the Orbetello Lagoon, Italy, and their characterization. J Appl Microbiol. 2007;103:2299–308.
24. Peña-Montenegro TD, Dussán J. Genome sequence and description of the heavy metal tolerant bacterium Lysinibacillus sphaericus strain OT4b.31. Stand Genomic Sci. 2013;9:42–56.
25. Khan AR, Park G, Asaf S, Hong S, Jung K, Shin J. Complete genome analysis of Serratia marcescens RSC-14: A plant growth-promoting bacterium that alleviates cadmium stress in host plants. PLoS One. 2017;1–17.
26. Jafarzade M, Mohamad S, Usup G, Ahmad A. Heavy metal tolerance and antibiotic susceptibility of red pigmented bacteria isolated from marine environment. Nat Resour. 2012;3:171–4.
27. Dharni S, Kumar A, Samad A, Dhar D. Impact of plant growth promoting Pseudomonas monteilii PsF84 and Pseudomonas plecoglossicida PsF610 on metal uptake and production of secondary metabolite (monoterpenes) by rose-scented geranium (Pelargonium graveolens cv . bourbon) grown on tannery sludg. Chemosphere [Internet]. Elsevier Ltd; 2014;117:433–9. Available from: http://dx.doi.org/10.1016/j.chemosphere.2014.08.001
28. Kumar K V, Singh N, Behl HM, Srivastava S. Influence of plant growth promoting bacteria and its mutant on heavy metal toxicity in Brassica juncea grown in fly ash amended soil. Chemosphere. 2008;72:678–83.
Recibido: 21 noviembre 2017
Aprobado: 15 enero2018
Manuel Alexander Rojas 1. Oscar Amable Vivanco Galván 2
Ecología y Evolución de Sistemas Microbianos. Departamento Ciencias Biológicas.
Universidad Técnica Particular de Loja. Ecuador
