2017.02.02.3
Files > Volume 2 > Vol 2 No 2 2017 > Investigaciones
INVESTIGATION / RESEARCH
Caracterización molecular y criopreservación de hongos y bacterias asociados a la rizósfera de especies vegetales acumuladoras de metales pesados en suelos contaminados por minería.
Molecular characterization and cryopreservation of fungi and bacteria associated to the rhizosphere of heavy metal accumulation vegetable species in soils contaminated by mining.
José Francisco Ochoa 1. Aminael Sánchez-Rodríguez 2.
Available from: http://dx.doi.org/10.21931/RB/2017.02.02.3
_______________________________________________________________________________________________________________________
RESUMEN
Los microorganismos son un claro ejemplo de la enorme biodiversidad que contiene el suelo. Se procedió al aislamiento in vitro, caracterización molecular e identificación de microorganismos de suelo de rizósfera asociados a Miconia zamorensis y Erato polymnoides, considerados como acumuladores de metales pesados en la zona minera de Chinapintza - Zamora Chinchipe. Las cepas se identificaron mediante la secuenciación del gen 16S ARN ribosomal en bacterias, y se identificó la presencia de géneros como Bacillus, Lysinibacillus y Serratia. En hongos se utilizó la secuencia del gen ITS ARN ribosomal y se identificaron los géneros Aspergillus, Talaromyces, Penicillium, Trichoderma, Nodulisporium, Fusarium, Rhizopus, Mucor, Umbelopsis e Hypochnicium. Tales géneros forman parte del suelo y se consideran como tolerantes a ecosistemas con elevada contaminación de metales pesados. Nuestros resultados serán el punto de partida de información sobre la potencialidad que brindan estos microorganismos para desarrollar estrategias de biorremediación en la zona de estudio. Debido a la gran demanda de elaborar colecciones de microorganismos con fines de biorremediación, se planteó la criopreservacion de los cultivos puros bacterianos y fúngicos. Se obtuvo una viabilidad efectiva en la reactivación de los cultivos bacterianos, mientras que en hongos la mayoría de cultivos fueron reactivados con éxito.
Palabras Clave: Microorganismos de suelo, aislamiento in vitro, criopreservación, caracterización molecular, 16S ARN ribosomal, ITS ARN ribosomal.
_______________________________________________________________________________________________________________________
ABSTRACT
Microorganisms are a clear example of the enormous biodiversity contained in the soil. It proceeded to the in vitro isolation, molecular characterization and identification of microorganism’s rhizosphere soil associated with Miconia zamorensis and Erato polymnoides, regarded as accumulators of heavy metals in the mining area of Chinapintza - Zamora Chinchipe. The strains were identified by sequencing the 16S ribosomal RNA gene in bacteria, and it was identified the presence of genera such as Bacillus, Serratia and Lysinibacillus. In fungi was used the sequence of ITS ribosomal RNA gene and it was identified the genres Aspergillus, Talaromyces, Penicillium, Trichoderma, Nodulisporium, Fusarium, Rhizopus, Mucor, Hypochnicium and Umbelopsis. Such genres form part of the soil and are considered as tolerant to ecosystems with heavy metals contamination. Our results will be the starting point of information on the potentiality of these microorganisms to develop bioremediation strategies in the study area. Due to the high demand to elaborate collections of microorganisms for bioremediation purposes, it was proposed the cryopreservation of bacterial and fungal pure cultures. It was obtained an effective viability in the reactivation of bacterial cultures, while in fungi most cultures were reactivated successfully.
Keywords: Soil microorganisms, in vitro isolation, cryopreservation, molecular characterization, 16S ribosomal RNA, ITS ribosomal RNA.
_______________________________________________________________________________________________________________________
INTRODUCCIÓN
En Ecuador la actividad minera desorganizada y artesanal genera un alto grado de contaminación ambiental, sobre todo la relacionada con la extracción de oro produciendo un vertimiento muy elevado de mercurio al suelo y riachuelos cercanos a las áreas de extracción (1). En la actualidad los ecosistemas contaminados están en el centro de atención tanto de gobernantes como de la comunidad científica y la creación de estrategias para recuperar y disminuir el impacto de la elevada contaminación por metales pesados es un activo campo de investigación (2).
El empleo de especies vegetales de por sí no es una estrategia de biorremediación con un balance costo/beneficio favorable (3). Una alternativa para mejorar estrategias de remediación basadas en plantas acumuladoras de metales pesados consiste en asociar las plantas con microorganismos del suelo (4). Estos microorganismos tienen la capacidad de facilitar el proceso de extracción de metales pesados por parte de la rizósfera de las plantas (5). La rizósfera es considerada como un “punto caliente” de las actividades microbianas, en donde las interacciones entre las raíces y los microorganismos constituyentes del suelo (minerales y materia orgánica) tienen lugar (5). En general, los microorganismos sirven como intermediarios entre la planta que requiere nutrientes o minerales solubles, y el suelo, que contiene los nutrientes necesarios pero a menudo en bajas concentraciones y formas inaccesibles para las plantas (6). De este modo los microorganismos de la rizósfera proporcionan un enlace crítico entre las plantas y el suelo (7). Por lo tanto, los microorganismos han desarrollado estrategias diversas que han propiciado el florecimiento de una serie de interacciones sinérgicas, tanto entre sí como con la planta (7).
Los microorganismos consumen metales pesados de forma activa (bioacumulación) o pasivamente (adsorción) (3). Por ello tienen la capacidad de inmovilizar metales por medio del secuestro o acumulación celular o a través de procesos de precipitación extracelular (8). Los efectos de los metales pesados sobre las comunidades microbianas pueden tener consecuencias dramáticas para el funcionamiento de todo el ecosistema (9). Si los metales se encuentran en concentraciones suficientemente altas pueden reducir el tamaño de las poblaciones microbianas, destruyendo sus estructuras y reduciendo sus actividades biológicas (10). Por consiguiente la estrecha interacción entre los microorganismos de la rizósfera y las plantas conducen a un aumento en las actividades relacionadas con la recuperación de suelos contaminados (11).
Basados en estos antecedentes, en el presente proyecto se decidió explorar la diversidad de comunidades microbianas asociadas a la rizósfera de especies vegetales acumuladoras de metales pesados en suelos contaminados, así como estrategias para su conservación en condiciones in vitro para facilitar aplicaciones de fitoremediación.
MATERIALES Y MÉTODOS
Aislamiento in vitro de cultivos microbianos.
Se realizó el aislamiento de microorganismos de suelo de rizósfera de Miconia zamorensis y Erato polymnoides identificadas como especies acumuladoras de metales pesados en la zona minera de Chinapintza – Zamora Chinchipe (Ecuador). Se establecieron dos transectos de 50m2, ubicados en las coordenadas 4°02'16.6"S 78°34'14.9"W. Transecto A: suelo contaminado al margen de un caudal de agua que sirve de vertedero de residuales de la actividad minera. Transecto C: área ubicada 300m al norte del transecto A.
Las cepas se aislaron utilizando la técnica de dilución en serie y fueron cultivadas en cajas Petri con medios de cultivo específicos para bacterias (BBL™ Trypticase™ Soy Agar -TSA) y hongos (BD Difco™ Potato Dextrose Agar - PDA).
Criopreservación y viabilidad de cepas bacterianas y fúngicas.
Los cultivos bacterianos y fúngicos puros fueron conservados en criotubos de almacenamiento y como crioprotectante se empleó el glicerol. Finalmente se procedió a colocar los criotubos en congelación a -80°C. Posteriormente fueron reactivadas al cabo de un periodo de tiempo en congelación. En bacterias fue después de 8 semanas mientras que en hongos fue en el lapso de 12 semanas. Tal proceso se realizó para medir la viabilidad del método de criopreservación empleado.
Caracterización molecular de cepas bacterianas y fúngicas.
Se procedió a la obtención de ADN genómico de las cepas bacterianas y fúngicas reactivadas empleando el kit de extracción UltraClean Microbial DNA Isolation kit (MoBio) siguiendo el protocolo del fabricante. Posteriormente se amplifico el ADN genómico mediante la técnica de Reacción en cadena de la polimerasa (PCR).
Las cepas bacterianas se identificaron utilizando las secuencias del gen 16S ARN ribosomal. Las muestras de ADN fueron obtenidas para cada colonia individual de cada cepa y la amplificación del gen 16S ARN ribosomal se llevó a cabo por PCR. La amplificación del ADN se realizó utilizando el GoTaq PCR Kit (PROMEGA) utilizando las condiciones propuestas por (12). La amplificación se realizó mediante el empleo de los siguientes primers: EUB338 (ACT CCT ACG GGA GGC AGC AG) y EUB518 (ATT ACC GCG GCT GCT GG) (12). Se empleó el termociclador (Applied Biosystem) y las condiciones fueron 94°C-9min por 1 ciclo; 94°C-45seg, 55°C-45seg, 72°C-45seg por 30 ciclos; 72°C-7min por 1 ciclo.
Las cepas fúngicas se identificaron utilizando las secuencias del gen ITS ARN ribosomal. La amplificación por PCR del ADN se realizó utilizando el GoTaq Pcr Kit (PROMEGA). La amplificación se realizó utilizando los primers ITS1F (TCC GTA GGT GAA CCT GCG G) y 5.8S (CGC TGC GTT CTT CAT CG) (12). Se empleó el termociclador (ProFlex™ PCR System) con las siguientes condiciones: 95°C-2min por 1 ciclo; 95°C-1min, 50°C-30seg, 72°C-30seg por 40 ciclos; 72°C-5min por 1 ciclo.
Los productos de PCR amplificados se separaron por electroforesis en geles de agarosa al 1%. Las muestras que obtuvieron resultados positivos en relación a la observación de bandas fueron purificadas empleando el protocolo Wizard® SV Gel and PCR Clean-Up System siguiendo el protocolo del fabricante. Se obtuvo un volumen final de 30 µl por cada muestra que fue utilizada para la secuenciación.
Secuenciación y análisis bioinformático de los fragmentos amplificados de los microorganismos.
Las muestras purificadas de productos de PCR se secuenciaron empleando el servicio de la compañía Macrogen, Korea (http://www.macrogen.com/kor/). Las secuencias bacterianas y fúngicas fueron analizadas utilizando la herramienta BLAST en la base de datos del NCBI GenBank (https://www.ncbi.nlm.nih.gov/) para encontrar secuencias estrechamente relacionadas. Sobre la base de máxima identidad fueron seleccionadas las primeras cinco secuencias y alineadas conjuntamente con las secuencias de bacterias y de igual manera para los hongos, para ello se empleó la herramienta MAFFT (Versión 7, estrategia G-INS-i). Para la construcción de árboles filogenéticos se utilizó el método de Máxima Verosimilitud con 1000 réplicas de bootstrap, empleando el software de análisis genéticos MEGA6. Por medio de este análisis fue posible la identificación molecular de los microorganismos presentes en la rizósfera de las especies vegetales asociados a las muestras de suelo.
RESULTADOS Y DISCUSIÓN
Criopreservación y viabilidad de microorganismos aislados.
Para realizar un congelamiento efectivo es necesario mantener los microorganismos bajo ausencia de agua, lo que resultara en un estado de latencia de las células vivas para inhibir sus actividades bioquímicas (13), proporcionando las condiciones necesarias para estabilizar a los microorganismos preservados inmovilizando o reduciendo el contenido de agua de las muestras almacenadas (14). Esto concuerda con los resultados obtenidos en la criopreservacion bacteriana debido a que se obtuvo un 100% de viabilidad en la reactivación de cada una de las muestras al cabo del periodo en congelación. Entre los principales factores que permitieron tal recuperación se encuentra que se aplicó como crioprotectante al glicerol. Según (15) el glicerol mediante el aumento de la concentración total de solutos reduce la cantidad de hielo formada, que es la causa del daño al momento de someter a microorganismos a extremas temperaturas de congelación, y el glicerol protege a las células contra este daño. La eficacia del glicerol como crioprotectante depende de una serie de propiedades: (a) es un compuesto soluble en agua y puede permanecer así a bajas temperaturas con el fin de producir una profunda depresión del punto de congelación; (b) capacidad de penetración celular; y (c) posee una baja toxicidad de modo que se puede utilizar a elevadas concentraciones (16).
En hongos de las 100 aislados sometidos a criopreservación únicamente fueron reactivados con éxito 90 muestras. Puede existir el caso que múltiples factores afecten la eficacia de la criopreservación que van desde procedimientos pre y post tratamiento. Estos incluyen, concentración del crioprotectante, fuentes de carbono y las intensidades de luz (17). Durante la fase de congelación, la formación de cristales de hielo se produce, y es letal cuando se forma intracelularmente, por lo tanto la prevención de dicha formación es altamente deseable (18). En el caso de hongos la criopreservacion fue efectiva porque un alto porcentaje de muestras fueron viables, pero hubo el caso de muestras que no lo hicieron. Por ello se pudo dar el caso de que varios de los factores mencionados afectaron los cultivos y no permitieron una viabilidad efectiva.
En el caso de hongos y bacterias se empleó para la criopreservación diferentes concentraciones de glicerol. Para hongos fue empleado al 10% y se obtuvo resultados satisfactorios en la congelación y posterior reactivación de las colonias fúngicas. A una concentración del 10%, el glicerol permite que la solución congelada sea bastante sólida a -80°C (19) y los hongos fueron cultivados en medio de agar previamente a ser almacenados, a esta concentración el glicerol cumple su función de evitar daños de congelación, tales como la formación de cristales de hielo (20). Por otro lado las colonias bacterianas fueron sometidas a concentraciones más altas de glicerol (80%), ya que a concentraciones mayores al 50% el cultivo puede permanecer parcialmente líquido (21) y las bacterias fueron criopreservadas en medio de cultivo líquido.
Identificación molecular bacteriana basado en la secuencia del gen 16S ARN ribosomal.
Un total de 27 secuencias de las muestras bacterianas fueron analizadas, las cuales fueron utilizadas para el análisis bioinformático. Se determinó que la mayoría de las secuencias presentaban una similitud mayor al 90% con las secuencias homologas más cercanas en las bases de datos del GenBank. La identificación filogenética de los aislados bacterianos basados en la secuencia parcial del gen 16S de ARN ribosomal se muestran en la Tabla 1. El análisis de las secuencias del gen 16S ARN ribosomal permitió identificar los siguientes géneros: Bacillus, Lysinibacillus y Serratia.

Tabla 1: Identificación molecular de aislados bacterianos.
Identificación molecular fúngica basado en la secuencia (ITS) de la región de ARN ribosomal.
Un total de 41 secuencias de las muestras fúngicas fueron analizadas, las cuales fueron utilizadas para el análisis bioinformático. Se determinó que la mayoría presentaban una similitud mayor al 90% con las secuencias homologas más cercanas en las bases de datos del GenBank. La identificación filogenética de los aislados fúngicos basados en la secuencia parcial del gen ITS de ARN ribosomal se muestran en la Tabla 2. El análisis de las secuencias del gen ITS ARN ribosomal permitió identificar los siguientes géneros: Aspergillus, Talaromyces, Penicillium, Trichoderma, Nodulisporium, Fusarium, Rhizopus, Mucor, Umbelopsis, e Hypochnicium.
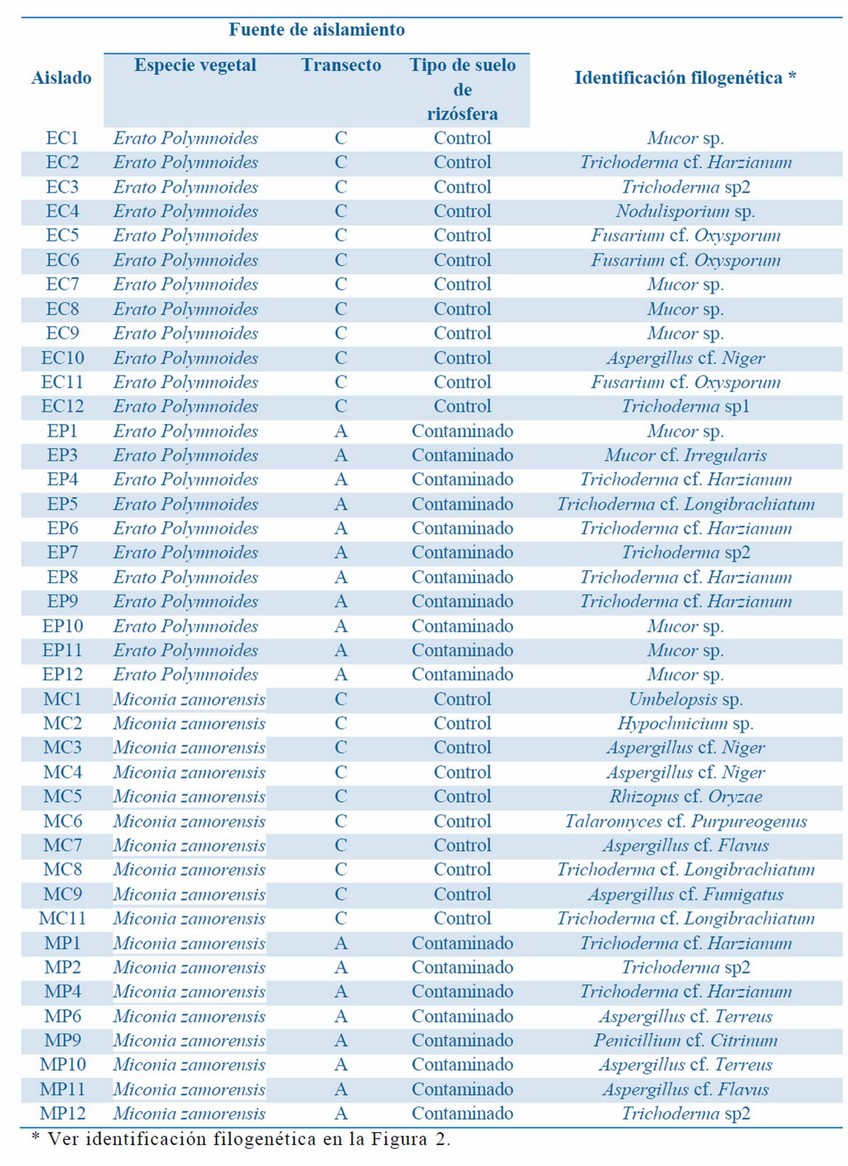
Tabla 2: Identificación molecular de aislados fúngicos.
Análisis filogenético de Bacterias.
La Figura 1 revela un árbol filogenético entre las cepas aisladas y las secuencias de varias especies de referencia del GenBank. Lo que ha permitido conocer las poblaciones de bacterias cultivables pertenecientes a las diferentes fuentes de asilamiento.
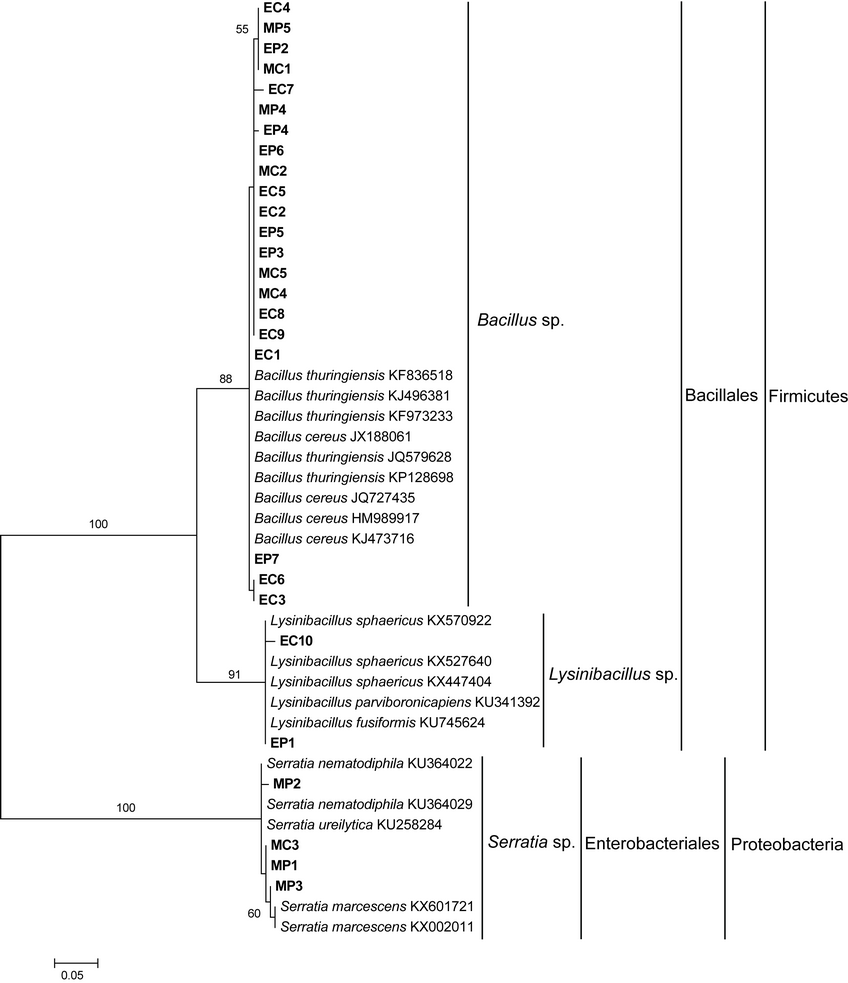
Figura 1: Árbol filogenético de Máxima Verosimilitud basado en la región 16S parcial de ARN ribosomal que muestra las relaciones filogenéticas de muestras bacterianas de los Transectos A y C. Solamente se muestran los valores de bootstrap superiores a 50%.
Se ha evidenciado la presencia de los géneros Bacillus, lysinibacillus y Serratia en los transectos A y C. Tal patrón de agregación se puede explicar porque que ambos transectos se encuentran en un área con un alto deterioro del medio ambiente. El transecto C pertenece a una zona minera abandonada, donde las actividades mineras se detuvieron cinco años antes del presente estudio. Por otro lado el transecto A es una zona intensiva de minería, en donde se están llevando a cabo una gran cantidad de actividades de minería de oro a pequeña escala. Otro de los motivos es que se determinó que en los tejidos (raíz, tallo y hojas) de Miconia Zamorensis y Erato polymnoides se encontraban considerables cantidad de Hg. En el transecto C una media de concentración de Hg de (0.4 ± 0.2 - mg kg-1), mientras que en el transecto A una media de concentración de Hg de (2.1 ± 1.8 - mg kg-1). Por lo tanto no existe gran diferencia en la concentración de Hg entre los transectos, lo que evidencia las similares condiciones de contaminación entre ambos sitios.
Es interesante indicar que los géneros dominantes detectados en las muestras de cada transecto se relacionan con cepas degradantes y resistentes a metales pesados, tales como Bacillus, Lysinibacillus y Serratia. (22) informaron que Bacillus es una cepa bacteriana de amplia resistencia al mercurio (Hg), entre las principales condiciones que permite tal resistencia se encuentra la esporulación, que es de carácter natural para favorecer el crecimiento de Bacillus bajo condiciones de estrés. Además (23) indicaron que bacterias formadoras de esporas resistentes a metales pesados como Bacillus pueden emplearse en sitios contaminados que permitan la biorremediación de ambientes tóxicos.
Las cepas del genero Lysinibacillus es ampliamente aplicada en la biorremediación de metales pesados (24), tales como Co, Cu, Cr y Pb (25). Otra de las cepas bacterianas identificadas pertenecen al género Serratia la cual ha sido identificada de naturaleza tolerante a metales (26). En un estudio realizado por (27) reportaron que Serratia posee un amplio rango de resistencia a múltiples metales pesados como Mn, Hg, Pb, Zn, Cr, Ni, Co.
Análisis filogenético de Hongos.
En la Figura 2 se muestra un árbol filogenético de las muestras fúngicas y las secuencias de varias especies de referencia del GenBank. Lo que ha permitido conocer las poblaciones de hongos cultivables pertenecientes a las diferentes fuentes de asilamiento.
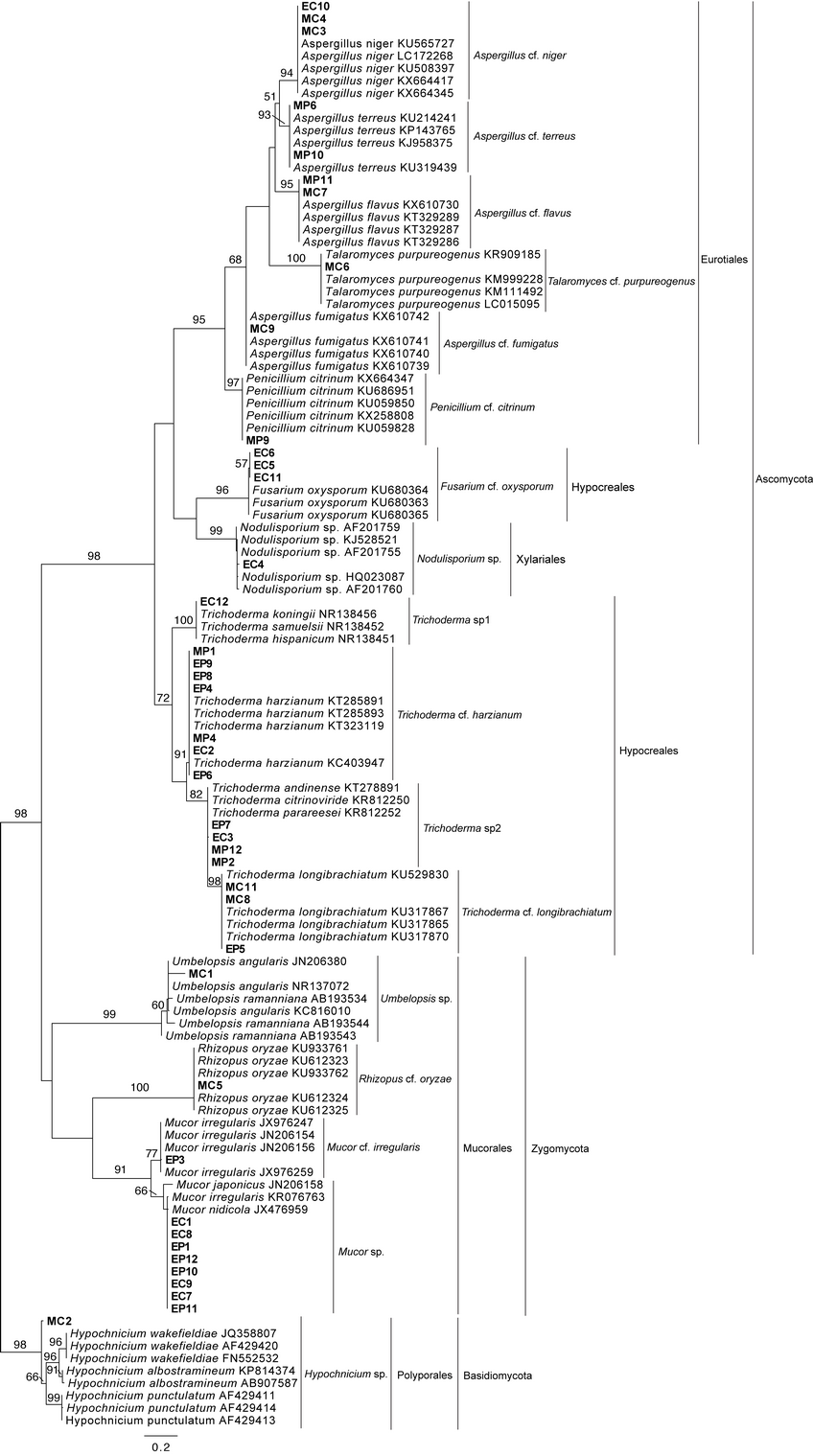
Figura 2: Árbol filogenético de Máxima Verosimilitud basado en la región ITS1 de ARN ribosomal que muestra las relaciones filogenéticas de muestras fúngicas de los Transectos A y C. Solamente se muestran los valores de bootstrap superiores a 50%.
En los transectos A y C se ha evidenciado que las concentraciones de Hg en el suelo se encuentran en cantidades significativas, por ello la mayoría de los géneros identificados en el presente estudio se encuentran tanto en los transectos A y C.
Los hongos son habitantes naturales del suelo y tienen una elevada potencialidad para la remediación en virtud de su enorme crecimiento, mayor biomasa, producción y extenso alcance de las hifas en el suelo (28). Son un grupo versátil, ya que pueden adaptarse y crecer en diversas condiciones extremas de pH, temperatura y disponibilidad de nutrientes, así como en ambientes con elevadas concentraciones de metales (29), como es el caso del área minera de Chinapintza, zona de la cual se aisló los hongos cultivables del suelo. Por otro lado ofrecen la ventaja de contener material en la pared celular que muestra excelentes propiedades de unión a los metales (30). Al describir la capacidad de crecer en altas concentraciones de metales, distinguimos hongos tolerantes a metales pesados los cuales se describen a continuación.
Especies del genero Aspergillus, Penicillium, Rhizopus y Trichoderma son únicos por su elevada tolerancia a metales y por su potencial de biosorción a diferentes metales pesados, además pertenecen a los microorganismos que son muy eficaces en la eliminación de metales pesados de ambientes contaminados (31). Por ello se han convertido en microorganismos dominantes en hábitats contaminados (32).
Otro de los géneros identificados fue Talaromyces el cual es tolerante a elevados niveles de Hg, Cu, Co, Pb y Cd (33). La adaptación a dichos metales fue el resultado de mecanismos fisiológicos y por procesos bioquímicos activados que confieren resistencia a varios metales pesados (34). También entre los géneros identificados se encuentran Nodulisporium el cual es un hongo endófito (35) y es eficaz como agente de control biológico contra una variedad de hongos patógenos (36); Mucor que ha sido aislado de suelos contaminados por metales pesados y se ha descrito una fuerte resistencia a Cd y Pb (37); Umbelopsis que es tolerante a elevadas concentraciones de Hg (38) e Hypochnicium que posee una elevada resistencia a Hg, As, Cd, Cu y Pb respectivamente (39). Finalmente entre los aislados se encontraron muestras pertenecientes al género Fusarium el cual puede desarrollarse en ambientes con presencia de Cr (40).
CONCLUSIONES
El presente estudio representa un primer avance en el reconocimiento de la microflora asociada a la rizósfera de las especies identificadas como acumuladoras, Miconia zamorensis y Erato polymnoides en la zona minera de Chinapintza. Se pudo obtener un alto nivel de diversidad con respecto a los géneros y especies de hongos y bacterias identificados, que serán el punto de partida de información sobre la potencialidad que brindan estos microorganismos para desarrollar estrategias de biorremediación en la zona de estudio. Se determinó la presencia de géneros bacterianos que pueden emplearse como agentes potenciales de biorremediación. Tales géneros bacterianos pueden tolerar ambientes contaminados con metales pesados debido a que poseen varias características metabólicas y catabólicas de resistencia. Por otro lado el conocimiento de la presente investigación proporcionará información acerca de los principales géneros fúngicos que se encuentran en la rizósfera de especies vegetales acumuladoras de metales pesados en Chinapintza, y con ello se pretenderá conocer acerca de la enorme diversidad de hongos que existe en el suelo.
La disponibilidad de materiales biológicos proporciona una ventaja significativa para establecer metodologías que permitan almacenar y preservar de forma estable durante determinados periodos de tiempo microorganismos basados en experimentación in vitro, y con ello apoyar programas de conservación, remediación de áreas contaminadas o el desarrollo de técnicas de preservación eficientes. La congelación a -80°C de las muestras microbianas con la metodología establecida en el estudio posibilitó el mantenimiento de la actividad enzimática y metabólica al cabo de un periodo de almacenamiento. Por lo tanto el método aplicado en la criopreservación de bacterias y hongos permitió obtener una viabilidad efectiva en la reactivación de las cepas puras.
AGRADECIMIENTOS
Con inmenso placer me gustaría dejar constancia de mi sincero agradecimiento al PhD. Aminael Sánchez, por su orientación, observaciones, críticas y estimulo durante el transcurro de este trabajo. También tengo una gratitud especial al Bq. Oscar Vivanco por su colaboración y las intervenciones oportunas en momentos cruciales. Expreso un profundo agradecimiento al Ing. Hernán Lucero por todos sus comentarios de apoyo y de motivación que extendió hacia mí durante el curso de la investigación. Finalmente a mi amiga y compañera Blga. Xiomara Recalde por su colaboración.
REFERENCIAS
1. Lovitz S. B. Scales of responsible gold mining: overcoming barriers to cleaner artisanal mining in southern Ecuador [Doctoral dissertation]. The University of Vermont; 2006.
2. Report I. Soil Contamination : Impacts on Human Health. Science for Environmental Policy. 2013; (5):1–29.
3. Dixit R, Wasiullah E, Malaviya D, Pandiyan K, Singh U, Sahu A, Shukla R & Paul, D. Bioremediation of Heavy Metals from Soil and Aquatic Environment: An Overview of Principles and Criteria of Fundamental Processes. Sustainability. 2015;7(2):2189-2212.
4. González Chávez M del C. Remediation of Soils Polluted with Heavy Metal Using Plants and Rhizospheric Microorganisms. Terra Latinoamericana. 2005;23(1):29–37.
5. Lambers H, Mougel C, Jaillard B, Hinsinger P. Plant-microbe-soil interactions in the rhizosphere: an evolutionary perspective. Plant and Soil. 2009;321(1-2):83-115.
6. Ortíz-Castro R, Contreras-Cornejo H, Macías-Rodríguez L, López-Bucio J. The role of microbial signals in plant growth and development. Plant Signaling & Behavior. 2009;4(8):701–12.
7. Mendes R, Garbeva P, Raaijmakers J. The rhizosphere microbiome: significance of plant beneficial, plant pathogenic, and human pathogenic microorganisms. FEMS microbiology reviews. 2013;37(5):634-663.
8. Wang Y, Shi J, Wang H, Lin Q, Chen X, Chen Y. The influence of soil heavy metals pollution on soil microbial biomass, enzyme activity, and community composition near a copper smelter. Ecotoxicology and Environmental Safety. 2007;67(1):75-81.
9. Turpeinen R. Interactions between metals, microbes and plants – Bioremediation of arsenic and lead contaminated soils. 1st ed. Lahti: University of Helsinki; 2002. 48.
10. Sobolev D, Begonia M. Effects of heavy metal contamination upon soil microbes: Lead-induced changes in general and denitrifying microbial communities as evidenced by molecular markers. International Journal of Environmental Research and Public Health. 2008;5(5):450–456.
11. Hongbo S, Liye C, Gang X, Kun Y, Lihua Z, Junna S. Progress in Phytoremediating Heavy-Metal Contaminated Soils. Detoxification of Heavy Metals. 2011. p. 73-90.
12. Fierer N, Jackson JA, Vilgalys R, Jackson RB. Assessment of Soil Microbial Community Structure by Use of Taxon-Specific Quantitative PCR Assays. Applied and environmental microbiology. 2005;71(7):4117–20.
13. Vekeman B, Hoefman S, De Vos P, Spieck E, Heylen K. A generally applicable cryopreservation method for nitrite-oxidizing bacteria. Systematic and Applied Microbiology. 2013;36(8):579–84.
14. Day J., Stacey G. Cryopreservation and freeze-drying protocols. 2da ed. Methods in molecular biology. Humana Press; 2007. 347 p.
15. Criste A, Giuburuncă M, Negrea O, Dan S, Marius Z. Research Concerning Use of Long-Term Preservation Techniques for Microorganisms. Animal Science and Biotechnologies. 2014;47(2):73–78.
16. Hubalek Z. Protectants used in the cryopreservation of microorganisms. Cryobiology. 2003;46(3):205–29.
17. Le Bui T. Cryopreservation, culture recovery and glucose induced programmed cell death in chlorophyte microalgae [Doctoral dissertation]. The University of Queensland; 2015.
18. Day J. Cryopreservation: Fundamentals, mechanisms of damage on freezing/thawing and application in culture collections. Nova Hedwigia. 2004;79(1–2):191–205.
19. Homolka L. Methods of Cryopreservation in Fungi. Laboratory Protocols in Fungal Biology. 2013;9–17.
20. Espinel-Ingroff A, Montero D, Martin-Mazuelos E. Long-Term Preservation of Fungal Isolates in Commercially Prepared Cryogenic Microbank Vials. Journal of Clinical Microbiology. 2004;42(3):1257–1259.
21. Lorv J, Rose D, Glick B. Bacterial Ice Crystal Controlling Proteins. Scientifica. 2014;2014:1–20.
22. Khare S, Ahmed N, Pant S, Das R. Characterization and evaluation of heavy metal tolerance of bacterial species from soil of waste area near Riyan steel rolling mills, Muzaffarnagar, India. Journal of Applied Natural Science. 2010;2:88–92.
23. Kumar A, Singh Bisht B, Datt Joshi V. Biosorption of Heavy Metals by four acclimated microbial species, Bacillus spp., Pseudomonas spp., Staphylococcus spp. and Aspergillus niger. Journal of Biological and Environmental Sciences. 2010;4(12):97–108.
24. Peña-Montenegro T, Dussán J. Genome sequence and description of the heavy metal tolerant bacterium Lysinibacillus sphaericus strain OT4b.31. Standards in Genomic Sciences. 2013;9(1):42–56.
25. Tuzen M, Dogan O, Usta C, Soylak M. Biosorption of copper ( II ), lead ( II ), iron ( III ) and cobalt ( II ) on Bacillus sphaericus -loaded Diaion SP-850 resin. Analytica Chimica Acta. 2007; 581(2):241-246.
26. Nageswaran N, Ramteke P, Verma O, Pandey A. Antibiotic Susceptibility and Heavy Metal Tolerance Pattern of Serratia Marcescens Isolated From Soil and Water. Biorremediation & Biodegradation. 2012;3(7):3–7.
27. Jafarzade M, Mohamad S, Usup G, Ahmad A. Heavy-metal tolerance and antibiotic susceptibility of red pigmented bacteria isolated from marine environment. Natural Resources. 2012;3(4):171–174.
28. Siddiquee S, Rovina K, Azad S, Naher L, Suryani S, Chaikaew P. Microbial & Biochemical technology heavy metal contaminants removal from wastewater using the potential filamentous fungi biomass : A Review. Microbial & Biochemical Technology. 2015;7(6):384–393.
29. Iram S, Zaman A, Iqbal Z, Shabbir R. Heavy metal tolerance of fungus isolated from soil contaminated with sewage and industrial wastewater. Pol J Environ Stud. 2013;22(3):691–697.
30. Khan A, Sharif M, Ali A, Noor S, Shah M, Mian I, & Ali, N. Potential of AM fungi in phytoremediation of heavy metals and effect on yield of wheat crop. American Journal of Plant Sciences. 2014;1578–1586.
31. Petková K, Jurkovic L, Simonovicova A, Cernansky S. Potential of Aspergilus niger in bioremediation of contaminated soils. International Multidisciplinary Scientific GeoConference: SGEM: Surveying Geology & Mining Ecology Management. 2013;1:757–762.
32. Iram S, Perveen K, Shuja N, Waqar K, Akhtar I, Ahmad I. Tolerance potential of different Species of Aspergillus as bioremediation tool - Comparative analysis. Journal of Microbiology. 2013;1(1):1–8.
33. Jaitly A. Mycoremediation : utilization of fungi for reclamation of heavy metals at their optimum remediation conditions. Biolife. 2015;3(1):77–106.
34. Romero M, Reinoso E, Urrutia M, Kiernan A. Biosorption of heavy metals by Talaromyces helicus: A trained fungus for copper and biphenyl detoxification. Electronic Journal of Biotechnology. 2006;9(3):221–226.
35. Nair D, Padmavathy S. Impact of endophytic microorganisms on plants, environment and humans. The Scientific World Journal. 2014;2014:11.
36. Porras-Alfaro A, Bayman P. Hidden Fungi , Emergent Properties : Endophytes and Microbiomes. Phytopathology. 2011;49(1):291.
37. Zhu S, Tang J, Zeng X, Wei B, Yang S, Huang B. Isolation of Mucor circinelloides Z4 and Mucor racemosus Z8 from heavy metal-contaminated soil and their potential in promoting phytoextraction with Guizhou oilseed rap. Journal of Central South University. 2015;22(1):88–94.
38. Crane S. Mercury effects on axeniclly grown fungal isolates and on pinus rigida and its ectomycorrhizal community [Doctoral dissertation]. The State University of New Jersey; 2011.
39. Kim M, Kim J, Kim G. Metal tolerance and sorption potential of indigenous basidiomycetous fungi. Division of Environmental Science & Ecological Engineering. 2011;88–89.
40. Khurshid S, Shoaib A, Javaid A, Abid K. Bioaccumulation of chromium by Fusarium oxysporum. ScienceAsia. 2016; 42(2):92-98.
Recibido: 10 abril 2017
Aprobado:15 mayo 2017
Aprobado:15 mayo 2017
José Francisco Ochoa 1. Aminael Sánchez-Rodríguez 2
1Departamento de Ciencias Biológicas. Universidad Técnica Particular de Loja. Ecuador
2MS2E Microbial Systems Ecology and Evolution / Ecología y Evolución de Sistemas Microbianos. Departamento de Ciencias Biológicas. Universidad Técnica Particular de Loja. Ecuador
Autor de correspondencia: [email protected]
